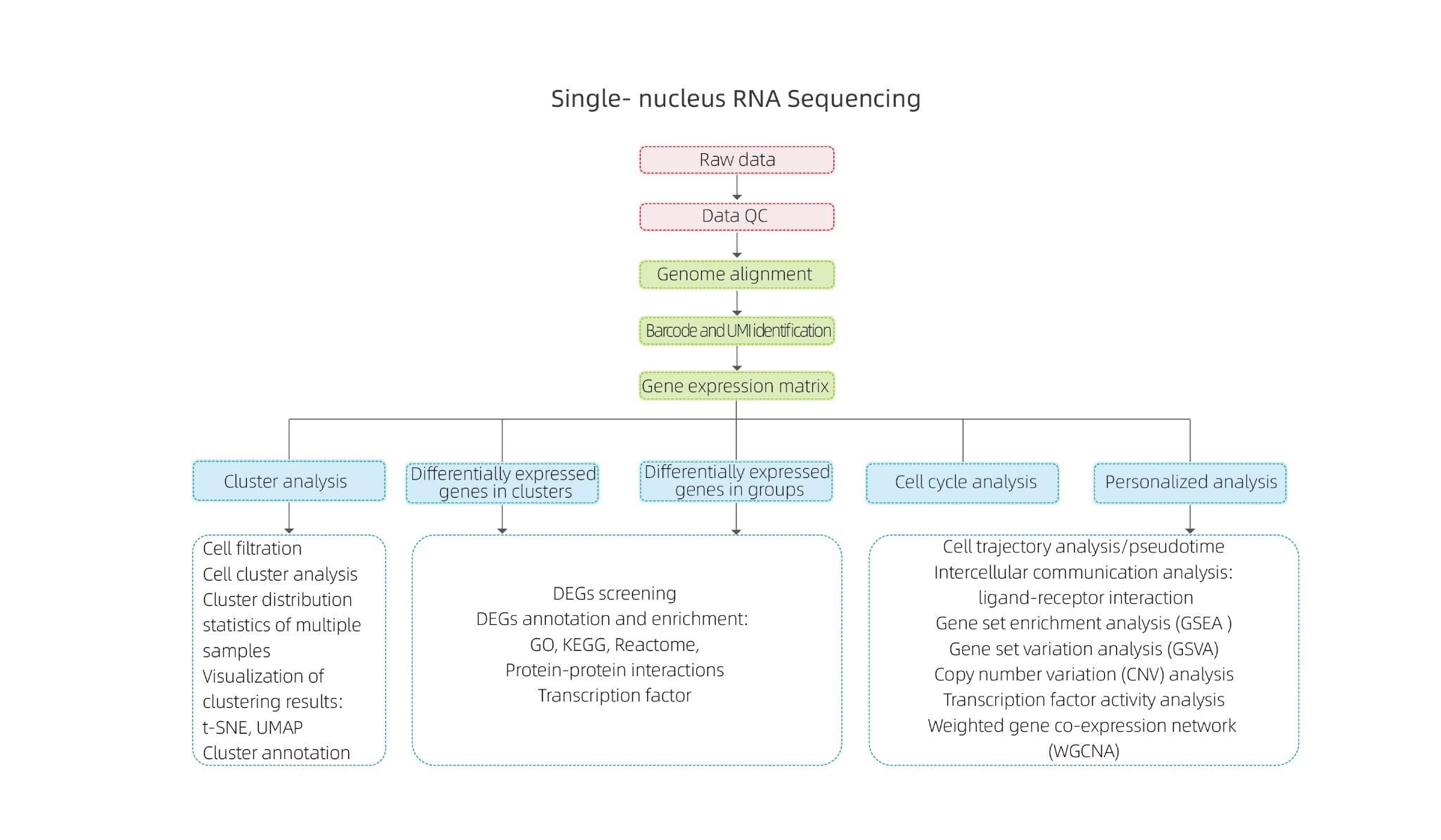
Einzelkern-RNA-Sequenzierung
Technisches Prinzip
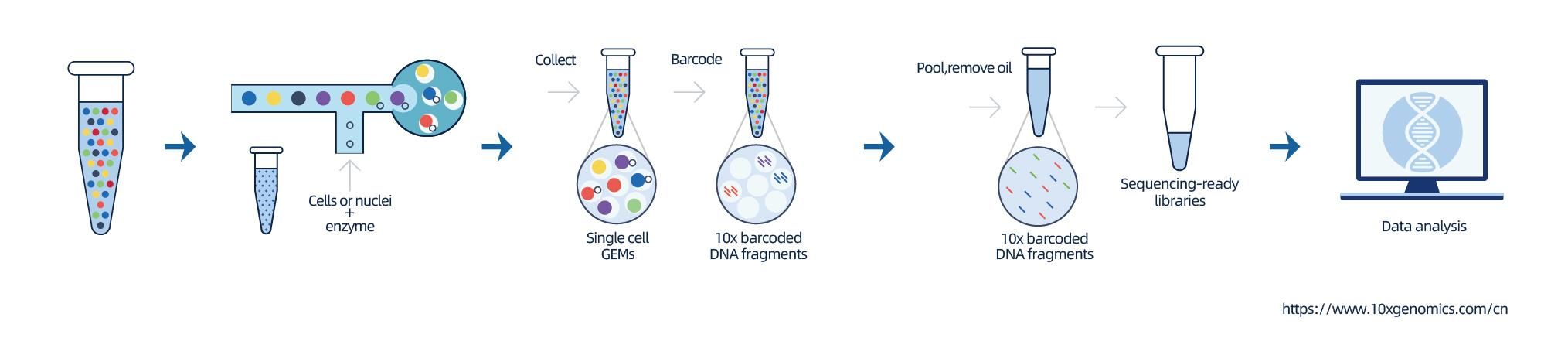
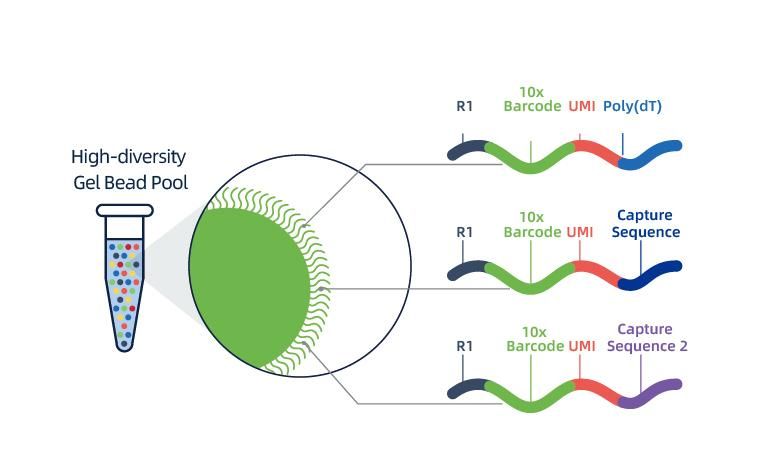
Die Isolierung der Kerne wird durch 10× Genomics ChromiumTM erreicht, das aus einem achtkanaligen Mikrofluidiksystem mit Doppelkreuzungen besteht.In diesem System werden Gelkügelchen mit Barcodes und Primer, Enzymen und einem einzelnen Kern in Öltropfen in Nanolitergröße eingekapselt, wodurch eine Gel Bead-in-Emulsion (GEM) entsteht.Sobald GEM gebildet sind, werden in jedem GEM eine Zelllyse und die Freigabe von Barcodes durchgeführt.mRNA wird mit 10-fachen Barcodes und UMI revers in cDNA-Moleküle transkribiert, die anschließend dem Aufbau einer Standard-Sequenzierungsbibliothek unterliegen.
Gewebe nicht für die Herstellung einer Einzelzellsuspension geeignet
| Zelle / Gewebe | Grund |
| Unfrisches gefrorenes Gewebe | Neue oder längst gespeicherte Organisationen können nicht abgerufen werden |
| Muskelzelle, Megakaryozyten, Fett… | Der Zelldurchmesser ist zu groß, um in das Instrument einzudringen |
| Leber… | Zu zerbrechlich zum Zerbrechen, unfähig, einzelne Zellen zu unterscheiden |
| Neuronenzelle, Gehirn… | Empfindlicher und leichter zu belasten, verändert die Sequenzierungsergebnisse |
| Bauchspeicheldrüse, Schilddrüse… | Reich an endogenen Enzymen, die die Produktion einer Einzelzellsuspension beeinflussen |
Einzelkern vs. Einzelzelle
| Einkernig | Einzelzelle |
| Unbegrenzter Zelldurchmesser | Zelldurchmesser: 10–40 μm |
| Das Material kann gefrorenes Gewebe sein | Das Material muss frisches Gewebe sein |
| Geringe Belastung durch gefrorene Zellen | Eine Enzymbehandlung kann eine Zellstressreaktion hervorrufen |
| Es müssen keine roten Blutkörperchen entfernt werden | Rote Blutkörperchen müssen entfernt werden |
| Nuklear drückt Bioinformationen aus | Die gesamte Zelle drückt Bioinformationen aus |
Leistungsbeschreibung
| Bibliothek | Sequenzierungsstrategie | Datenvolumen | Beispielanforderungen | Gewebe |
| 10× Genomics-Einzelkernbibliothek | 10x Genomics -Illumina PE150 | ca. 100.000 Lesevorgänge/Zelle.100–200 GB | Zellzahl: >2× 105 Zellkonz.bei 700–1.200 Zellen/μL | ≥ 200 mg |
Für weitere Einzelheiten zur Anleitung zur Probenvorbereitung und zum Service-Workflow wenden Sie sich bitte an aBMKGENE-Experte
Service-Workflow

Experimentdesign

Musterlieferung
Kernisolierung

Bibliotheksbau

Sequenzierung

Datenanalyse

Kundendienst
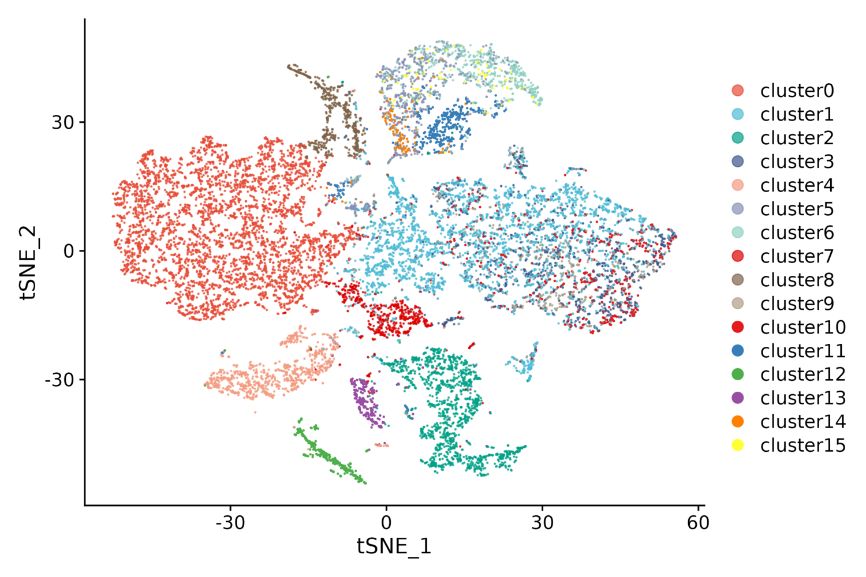
1. Spot-Clustering
2. Marker-Ausdruckshäufigkeits-Clustering-Heatmap
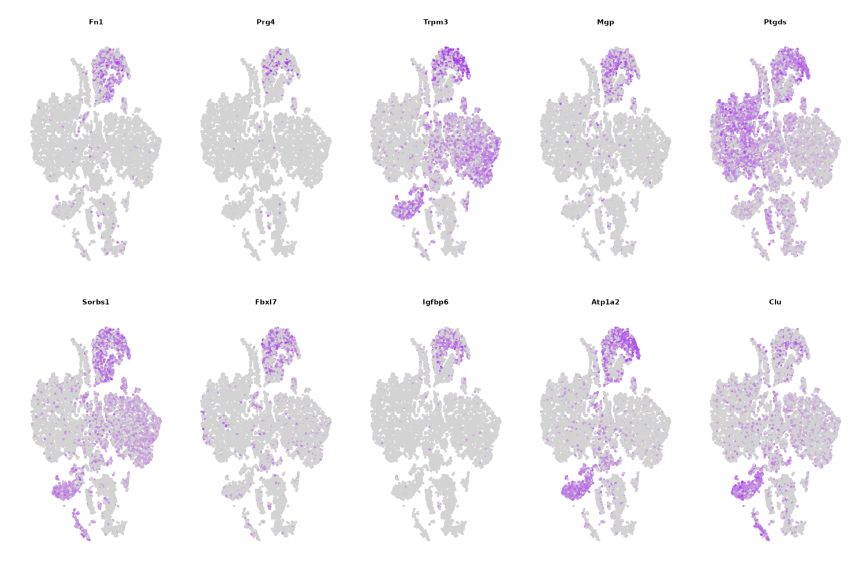
 3. Maker-Genverteilung in verschiedenen Clustern
3. Maker-Genverteilung in verschiedenen Clustern
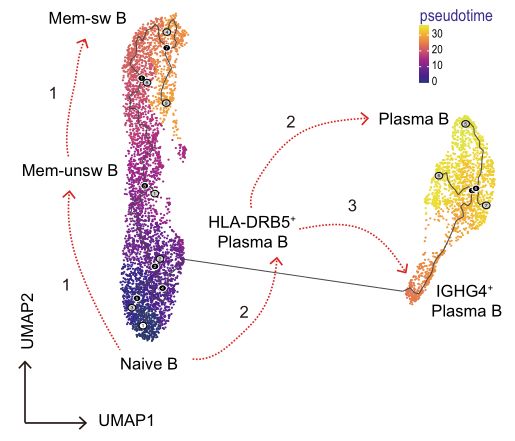
4.Zelltrajektorienanalyse/Pseudozeit