Bisulfit-Sequenzierung mit reduzierter Repräsentation (RRBS)
Leistungsbeschreibung
Plattform: NovaSeq-Plattform, PE150
Bibliothekstyp: 200–400 bp Insert-Bisulfit-behandelte DNA-Bibliothek
Sequenzierungsstrategie: Paired-End 150 bp
Empfohlene Datenausgabe: 10 GB Rohdaten/Probe
Beispielanforderungen
DNA-Menge: ≥ 2ug
DNA-Konzentration: ≥ 20 ng/μl.
Reinheit: OD260/280 = 1,8 bis 2,0 ohne Abbau oder RNA-Kontamination
Bioinformatische Analyse
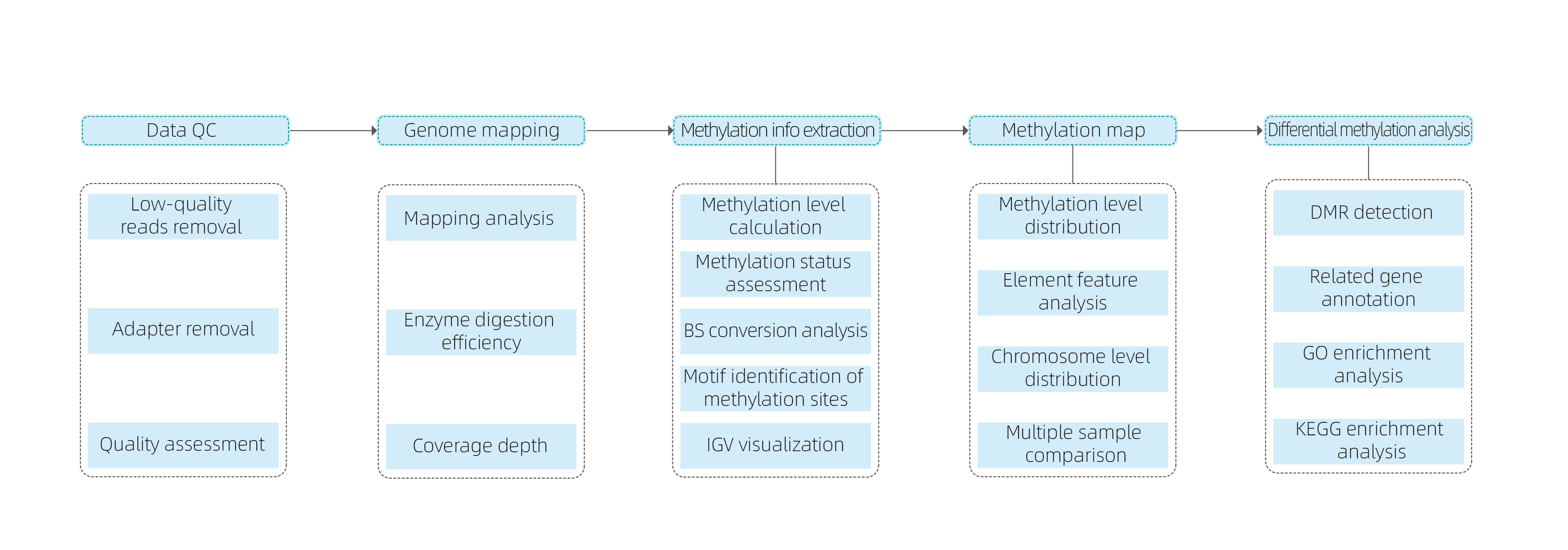
Service-Workflow

Musterlieferung

Bibliotheksbau

Sequenzierung

Datenanalyse
Kundendienst
a.Referenzstatistiken zur Genomausrichtung
Statistik der Mapping-Lesevorgänge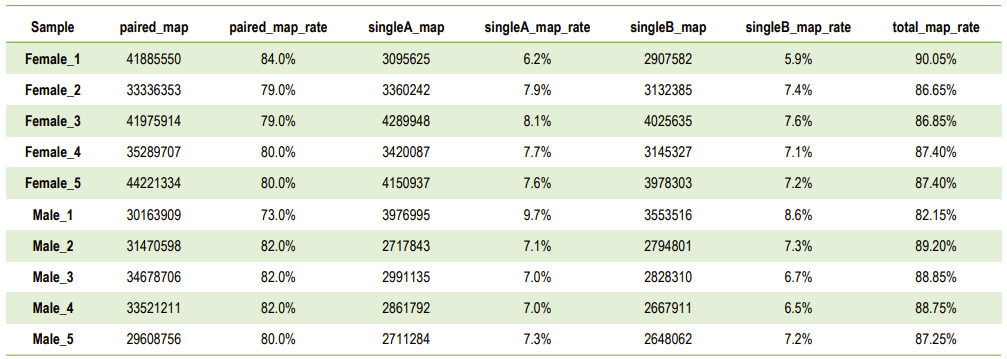
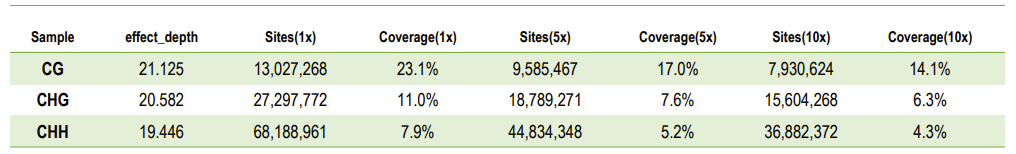
SStatistiken zur Sequenzierungstiefe und Abdeckung der C-Stelle
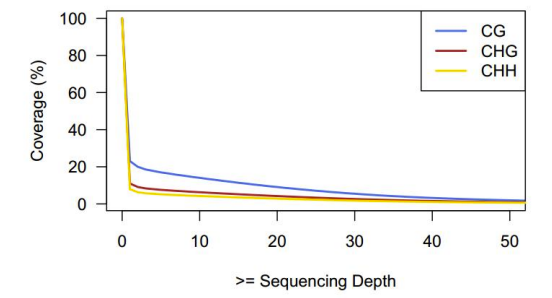
Akumulierte Deckung
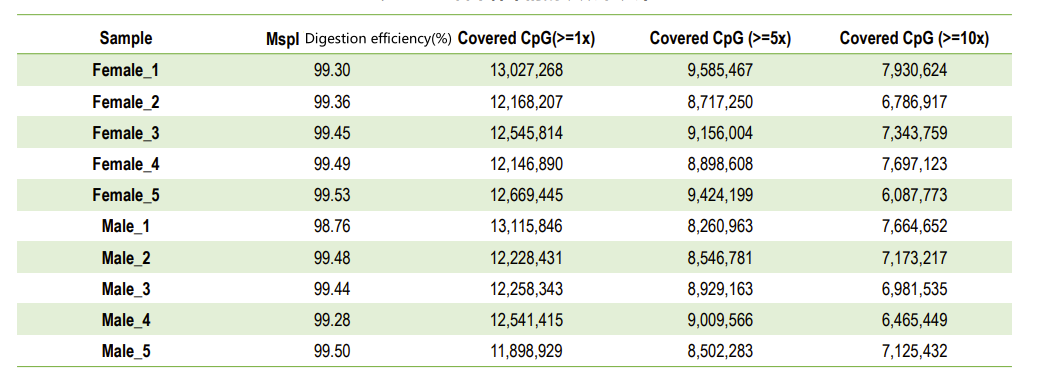
DVerdauungseffizienz jeder Probe
b.Nachweis der Methylierungsstelle
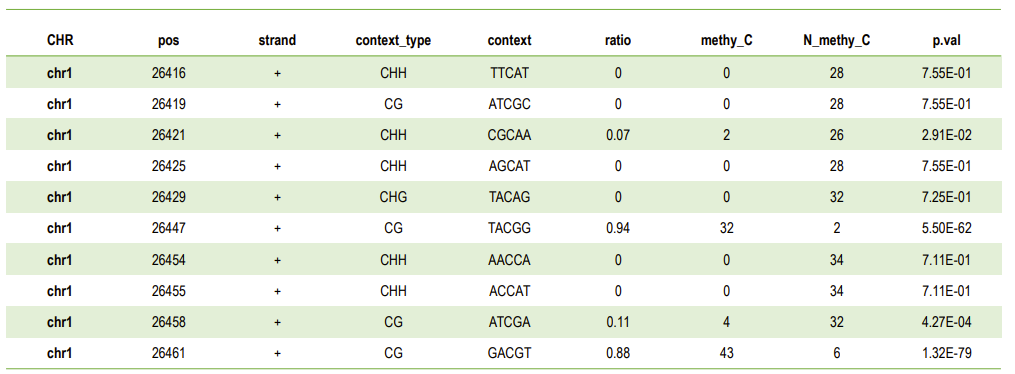
Liste der Methylierungsgrade an der C-Stelle
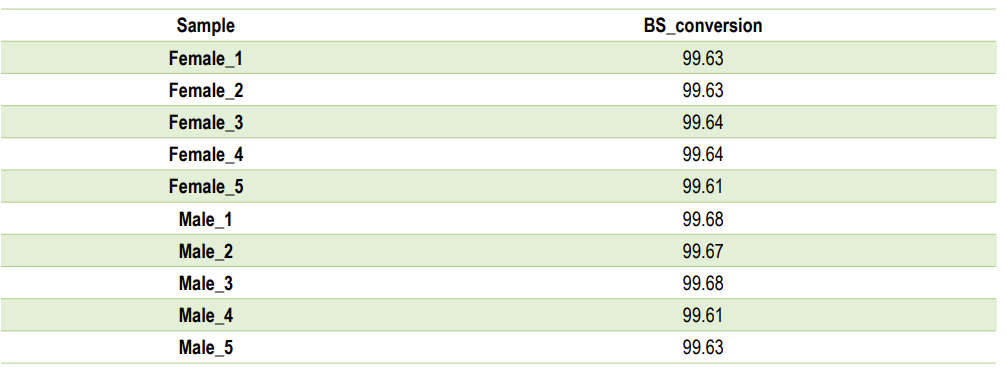
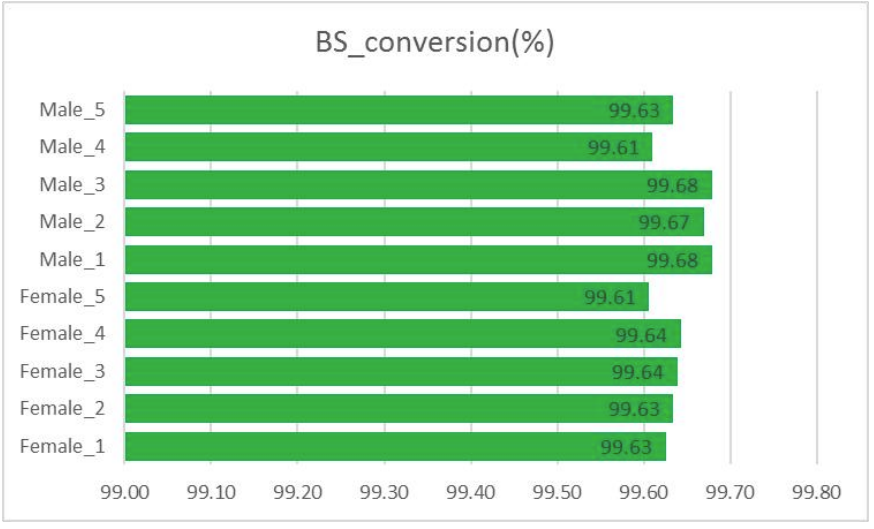
BS-Umbau
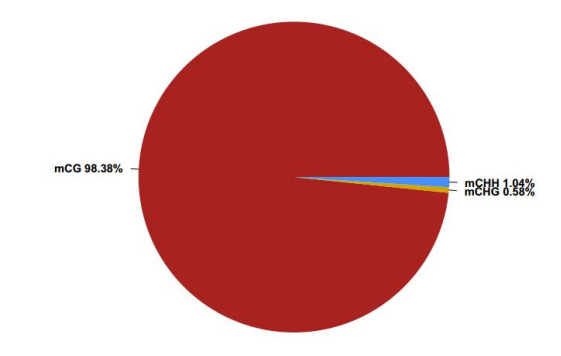
Verteilung des Methylierungsgrads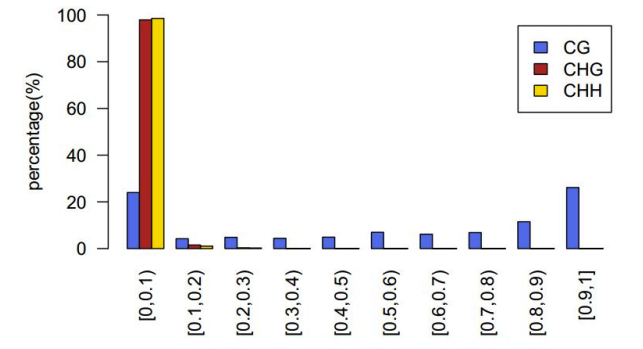
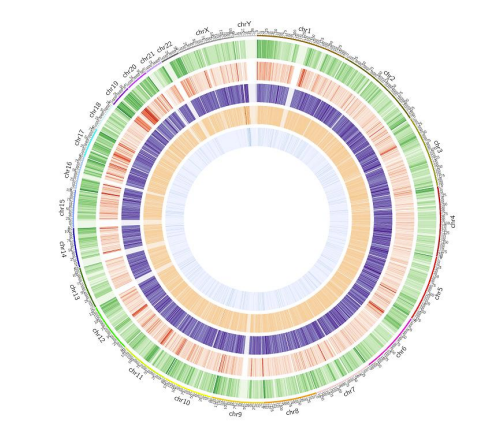
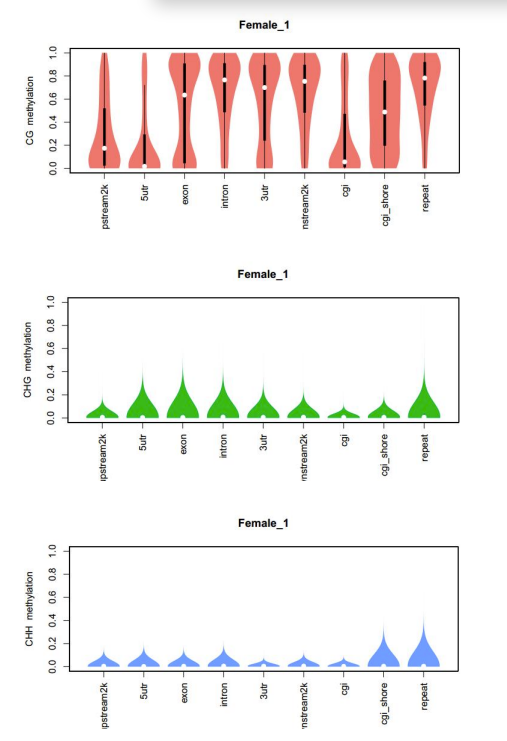
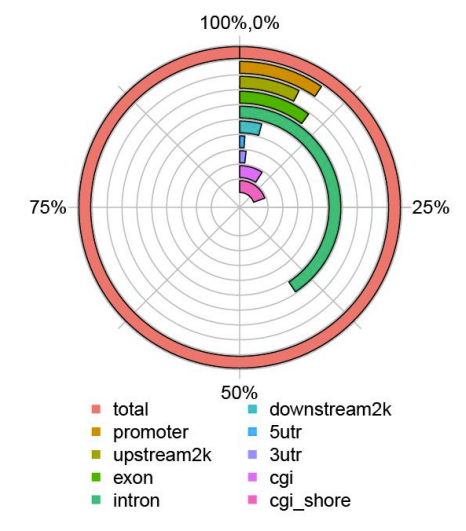
c.Methylierungskarte
GEnome-Element
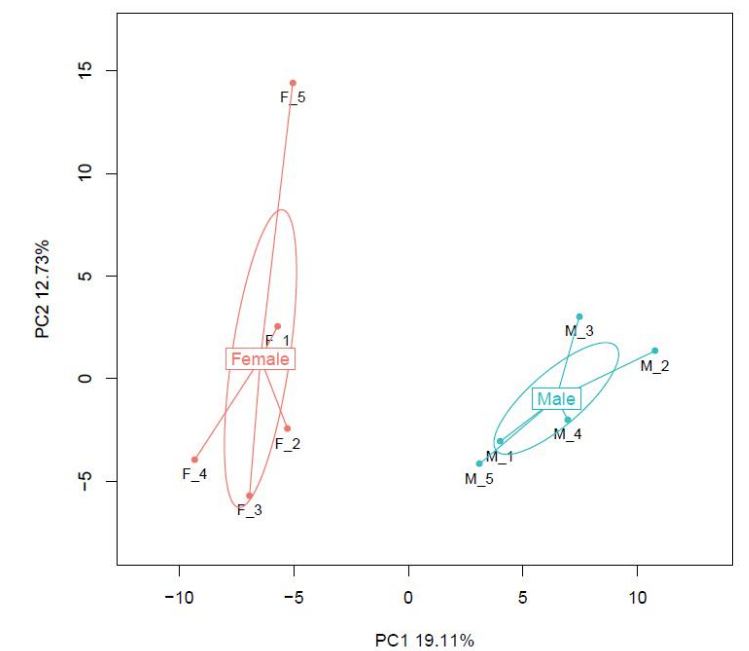
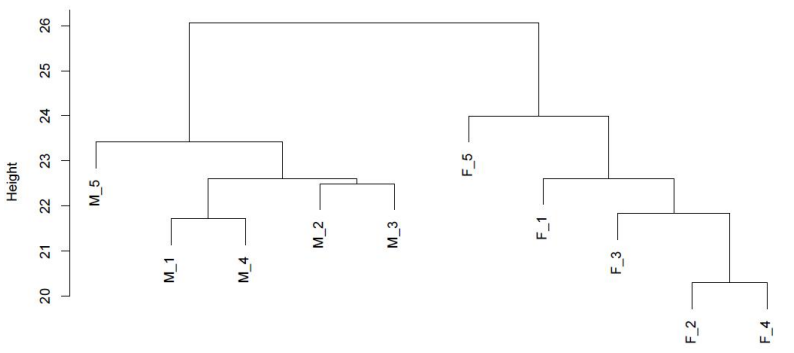
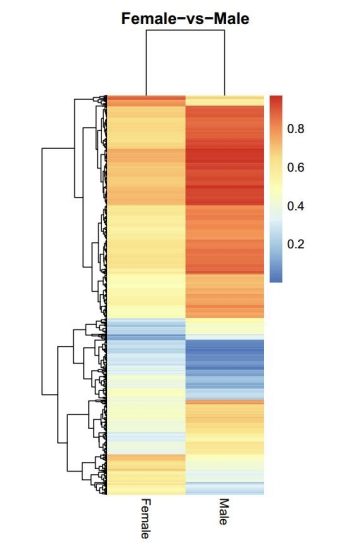
d.Vergleich der Methylierungsgrade zwischen Proben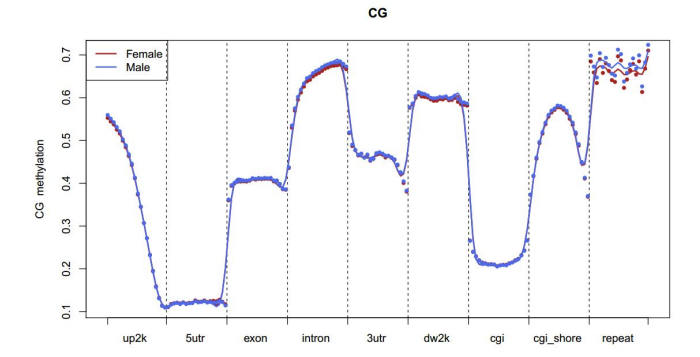
PAirwise Korrelation der CG-Methylierung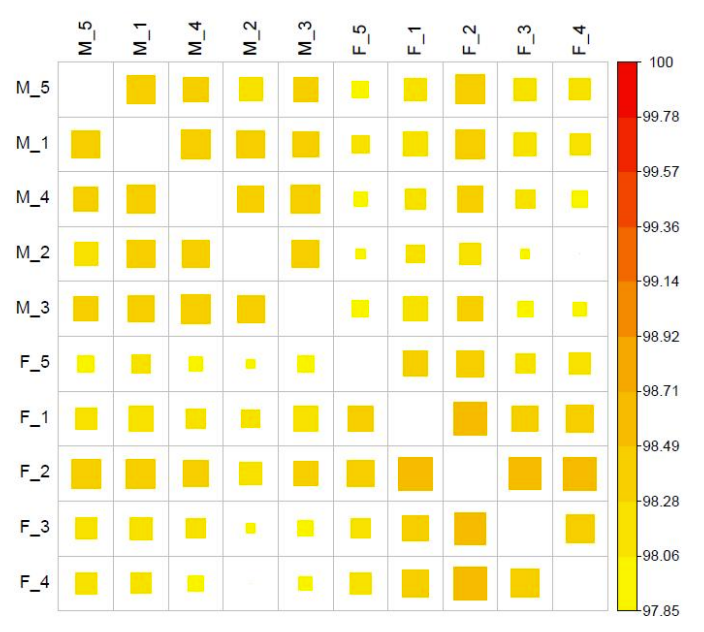
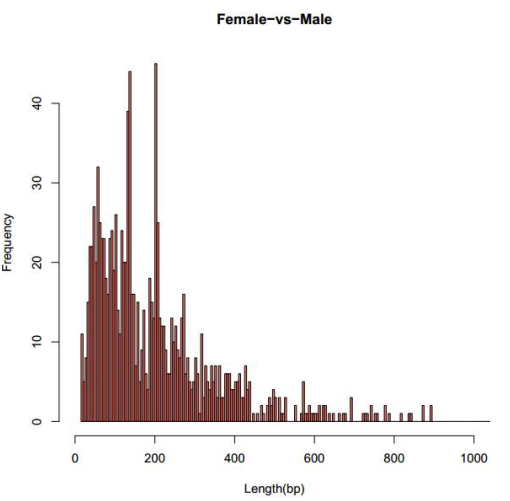
d.Differentielle Methylierungsanalyse
SStatistiken von DMR