
Heatmap
Die Matrixdatendatei wird zum Zeichnen von Heatmaps verwendet, mit denen Matrixdaten gefiltert, normalisiert und gruppiert werden können.Es wird hauptsächlich zur Clusteranalyse des Genexpressionsniveaus zwischen verschiedenen Proben verwendet.
Gen-Annotation
Die Annotation von Genfunktionen erfolgt durch Zuordnung von Sequenzen in der FASTA-Datei zu verschiedenen Datenbanken.

Gen-Annotation
Grundlegendes Suchtool für lokale Ausrichtung

CDS_UTR_Prediction
Dieses Tool wurde entwickelt, um kodierende Regionen (CDS) und nicht-kodierende Regionen (UTR) in bestimmten Transkriptsequenzen vorherzusagen, basierend auf dem Abgleich mit bekannten Proteindatenbanken und der ORF-Vorhersage.
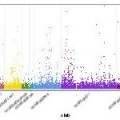
Manhattan-Grundstück
Der Manhattan-Plot ermöglicht die Anzeige von Daten mit einer großen Anzahl von Datenpunkten.Es wird häufig in genomweiten Assoziationsstudien (GWAS) verwendet.

Zirkus
Das CIRCOS-Diagramm ermöglicht die direkte Darstellung der SNP-, InDeL-, SV- und CNV-Verteilungen im Genom.

GO_Enrichment
TopGO ist ein Tool zur funktionalen Bereicherung.Das TopGO-Bioconductor-Paket besteht aus einer Differentialexpressionsanalyse, einer GO-Anreicherungsanalyse und einer Visualisierung der Ergebnisse.Es wird ein Ordner mit der Ausgabe namens „Graph“ generiert, der Ergebnisse für topGO_BP, topGO_CC und topGO_MF enthält.

WGCNA
WGCNA ist eine weit verbreitete Data-Mining-Methode zur Entdeckung von Gen-Koexpressionsmodulen.Es ist auf verschiedene Expressionsdatensätze anwendbar, einschließlich Microarray-Daten und Genexpressionsdaten, die aus der Sequenzierung der nächsten Generation stammen.

InterProScan
InterPro-Proteinsequenzanalyse und -klassifizierung

GO_KEGG_Enrichment
Dieses Tool dient zur Erstellung eines GO-Anreicherungshistogramms, eines KEGG-Anreicherungshistogramms und eines KEGG-Anreicherungspfads basierend auf dem bereitgestellten Gensatz und der entsprechenden Anmerkung.
