GENOM

Zusammenstellung und Analyse des Genoms der Biomassepflanze Miscanthus lutarioriparius im Chromosomenmaßstab
Nanoporensequenzierung |Illumina |Hi-C |RNA-Sequenzierung |Phylogenie
In dieser Studie leistete Biomarker Technologies technische Unterstützung bei der Nanoporensequenzierung, der De-novo-Genomassemblierung, der Hi-C-unterstützten Assemblierung usw.
Abstrakt
Miscanthus, eine rhizomatöse mehrjährige Pflanze, hat aufgrund ihrer hohen Biomasse und Stresstoleranz ein großes Potenzial für die Bioenergieproduktion.Wir berichten über eine Anordnung im ChromosomenmaßstabMiscanthus lutarioripariusGenom durch die Kombination von Oxford Nanopore-Sequenzierung und Hi-C-Technologien.Die 2,07-GB-Assembly deckt 96,64 % des Genoms ab, mit Contig N50 von 1,71 MB.Die Zentromer- und Telomersequenzen werden für alle 19 Chromosomen bzw. Chromosom 10 zusammengestellt.Der allotetraploide Ursprung von M. lutarioriparius wird mithilfe zentromerer Satellitenwiederholungen bestätigt.Die tetraploide Genomstruktur und mehrere chromosomale Umlagerungen im Vergleich zu Sorghum werden deutlich gezeigt.Tandem-Duplikat-Gene vonM. lutarioripariussind funktionell angereichert, nicht nur im Hinblick auf die Stressreaktion, sondern auch im Hinblick auf die Zellwandbiosynthese.Genfamilien im Zusammenhang mit Krankheitsresistenz, Zellwandbiosynthese und Metallionentransport werden stark erweitert und weiterentwickelt.Die Erweiterung dieser Familien könnte eine wichtige genomische Grundlage für die Verbesserung bemerkenswerter Merkmale von seinM. lutarioriparius.
Wichtige Statistiken zur Genomassemblierung
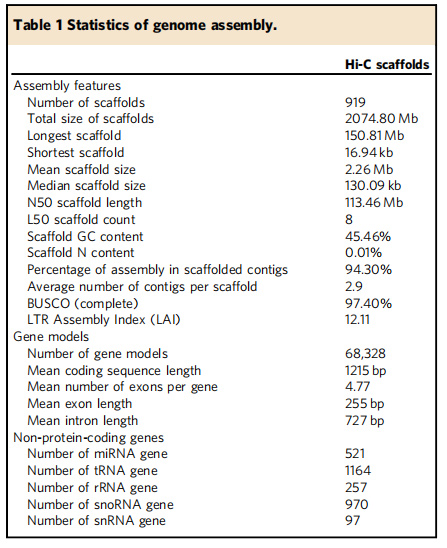
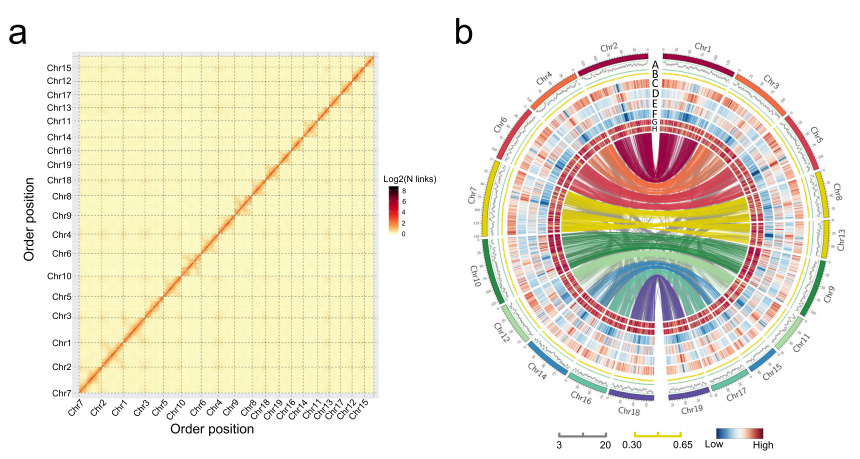
Figur.Überblick über die Genomassemblierung von M. lutarioriparius
Neuigkeiten und Highlights Ziel ist es, die neuesten erfolgreichen Fälle mit Biomarker-Technologien zu teilen und neue wissenschaftliche Errungenschaften sowie herausragende Techniken, die während der Studie angewendet wurden, zu erfassen.
Zeitpunkt der Veröffentlichung: 05.01.2022

