Sequenzierung des gesamten menschlichen Exoms
Servicevorteile
● Gezielte proteinkodierende Region: Durch die Erfassung und Sequenzierung der proteinkodierenden Region wird hWES verwendet, um Varianten im Zusammenhang mit der Proteinstruktur aufzudecken;
● Hohe Genauigkeit: Mit der hohen Sequenzierungstiefe erleichtert hWES die Erkennung häufiger Varianten und seltener Varianten mit Häufigkeiten von weniger als 1 %;
● Kostengünstig: hWES liefert etwa 85 % der Mutationen menschlicher Krankheiten aus 1 % des menschlichen Genoms;
● Fünf strenge QC-Verfahren, die den gesamten Prozess abdecken, mit garantiertem Q30>85 %.
Beispielspezifikationen
| Plattform
| Bibliothek
| Exon-Capture-Strategie
| Empfehlen Sie eine Sequenzierungsstrategie
|
|
Illumina NovaSeq-Plattform
| PE150 | Agilent SureSelect Human All Exon V6 IDT xGen Exome Hyb Panel V2 | 5 GB 10 GB |
Beispielanforderungen
| Beispielstyp
| Menge(Qubit®)
| Volumen
| Konzentration
| Reinheit (NanoDrop™) |
|
Genomische DNA
| ≥ 300 ng | ≥ 15 μL | ≥ 20 ng/μL | OD260/280=1,8-2,0 Keine Verschlechterung, keine Kontamination
|
Empfohlene Sequenzierungstiefe
Für Mendelsche Störungen/seltene Erkrankungen: effektive Sequenzierungstiefe über 50×
Für Tumorproben: effektive Sequenzierungstiefe über 100×
Bioinformatik


Service-Workflow

Musterlieferung

DNA-Extraktion

Bibliotheksbau

Sequenzierung

Datenanalyse

Datenlieferung

Kundendienst
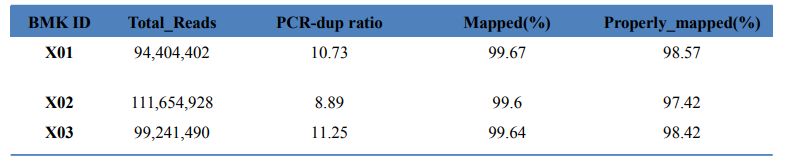
1. Ausrichtungsstatistik
Tabelle 1 Statistik des Kartenergebnisses
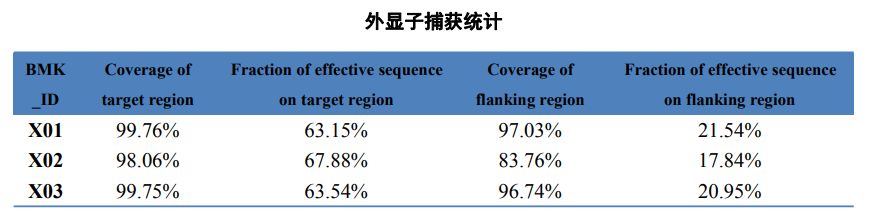
Tabelle 2 Statistik der Exom-Erfassung
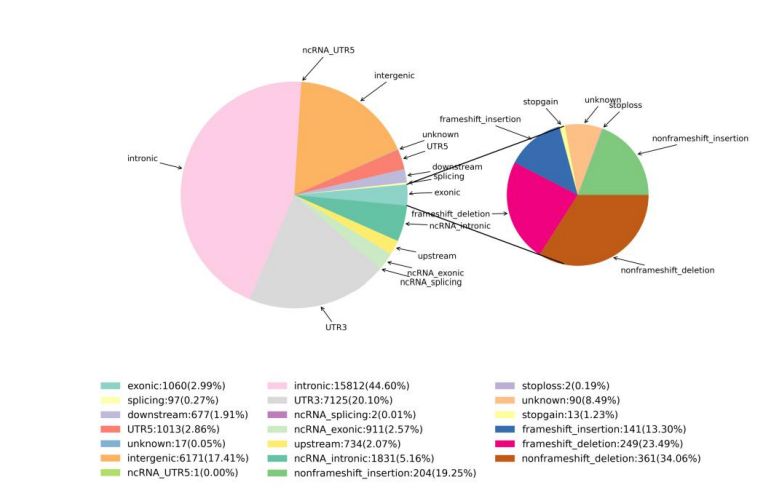
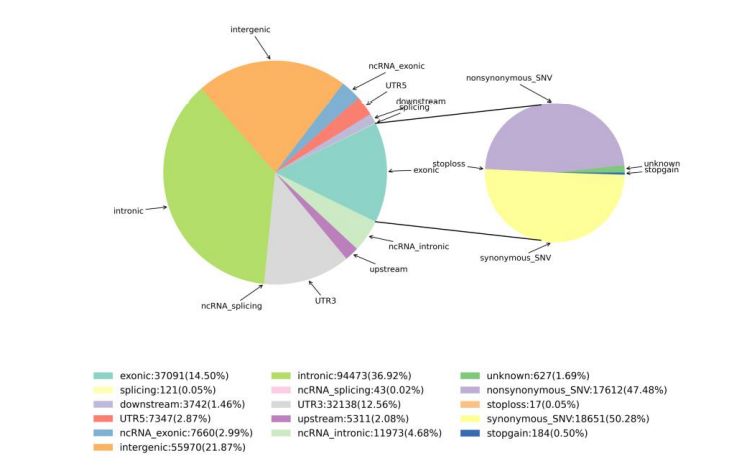
2. Variationserkennung
Abbildung 1 Statistik von SNV und InDel
3. Erweiterte Analyse
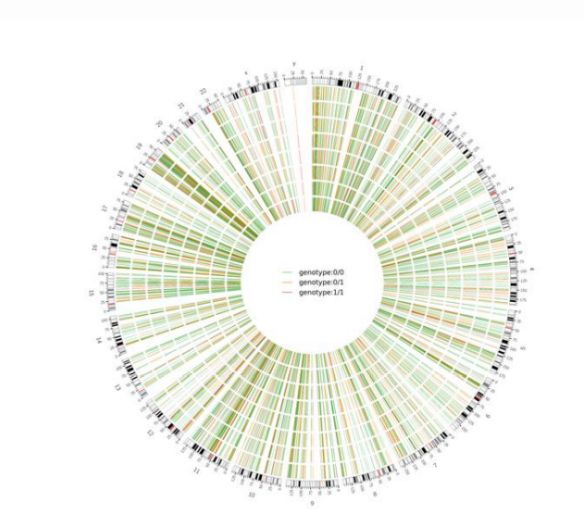
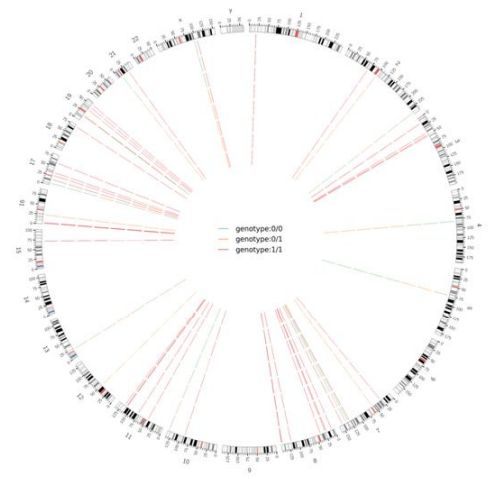
Abbildung 2 Circos-Diagramm von genomweit schädlichem SNV und InDel
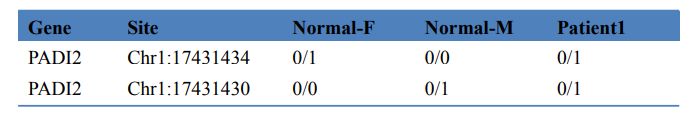
Tabelle 3 Screening krankheitsverursachender Gene