BMKMANU S1000 räumliches Transkriptom
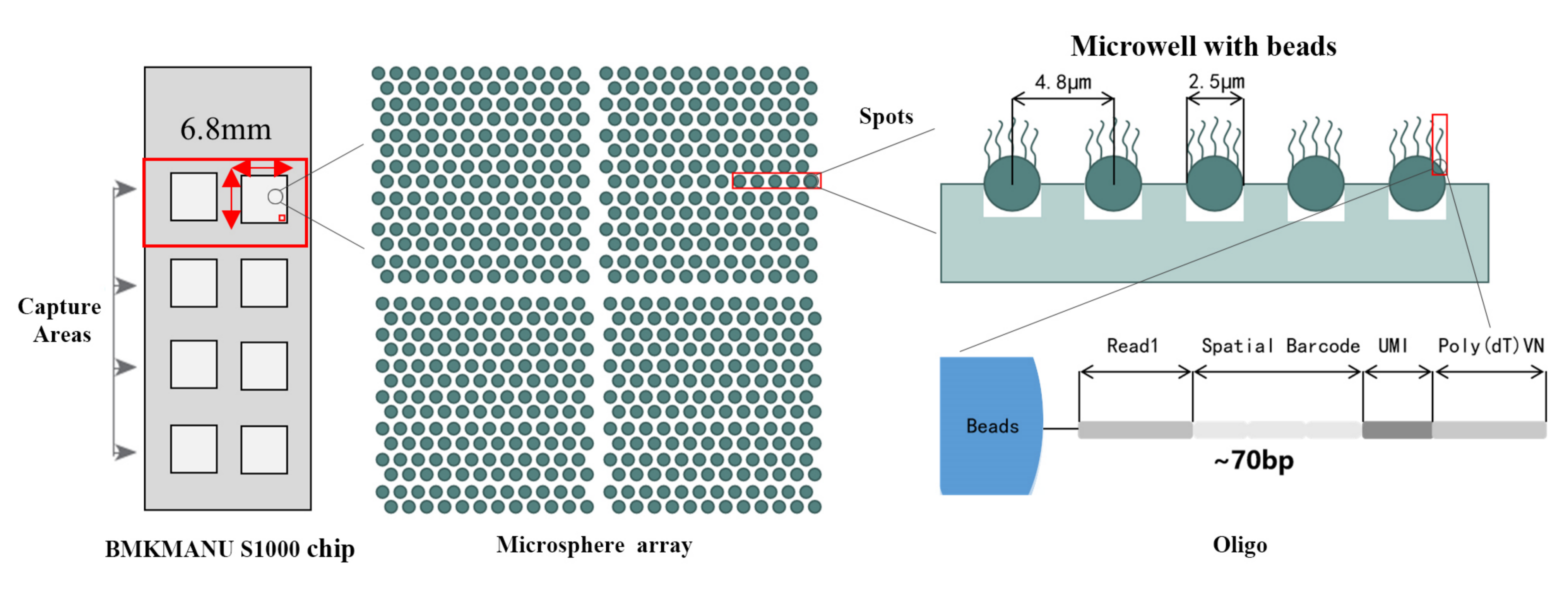
Technisches Schema des räumlichen Transkriptoms BMKMANU S1000
Vorteile von BMKMANU S1000
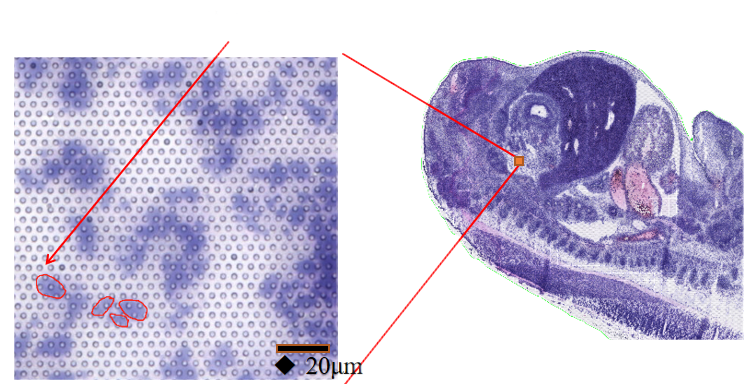
1) Subzelluläre Auflösung: Jeder Erfassungsbereich enthielt >2 Millionen räumliche Barcode-Spots mit einem Durchmesser von 2,5 µm und einem Abstand von 5 µm zwischen den Spotzentren, was eine räumliche Transkriptomanalyse mit subzellulärer Auflösung (5 µm) ermöglichte.
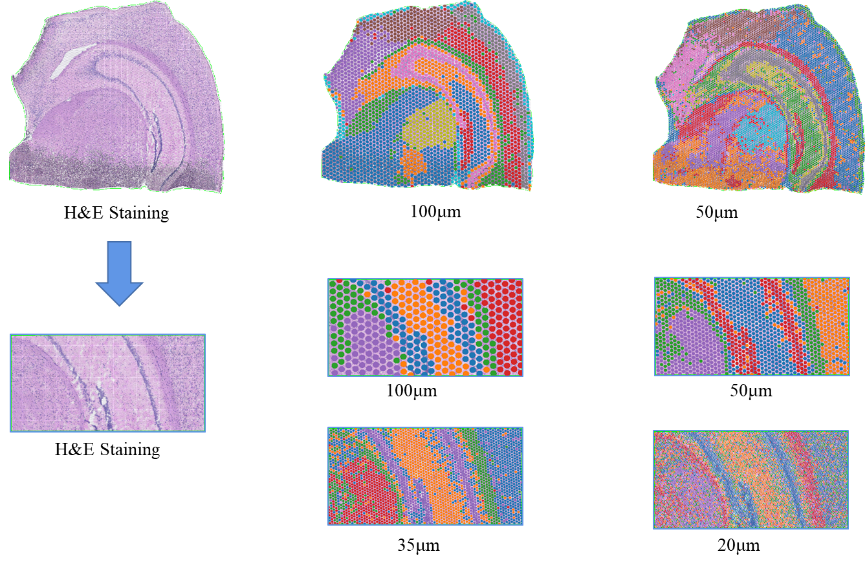
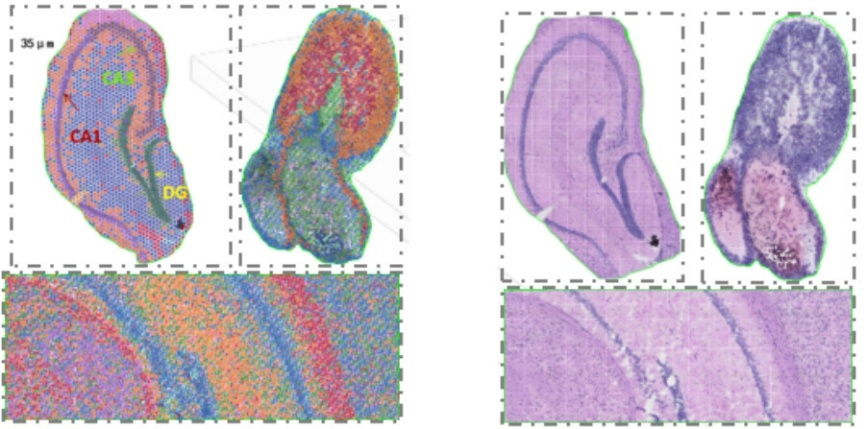
2) Mehrstufige Auflösungsanalyse: Flexible mehrstufige Analyse im Bereich von 100 μm bis 5 μm, um verschiedene Gewebemerkmale mit optimaler Auflösung aufzulösen.
3) Umfassende Transkriptom-Profilierung: Vom gesamten Gewebeobjektträger erfasste Transkripte können ohne Einschränkung der Anzahl der Zielgene und des Zielbereichs analysiert werden.
Leistungsbeschreibung
| Bibliothek | Sequenzierungsstrategie | Datenausgabe empfohlen |
| S1000-cDNA-Bibliothek | BMKMANU S1000-Illumina PE150 | 60 GB/Probe |
Probenanforderungen
| Probe | Nummer | Größe | RNA-Qualität |
| OCT-eingebetteter Gewebeblock | 2-3 Blöcke/Probe | Ca.6,8x6,8x6,8 mm3 | RIN≥7 |
Für weitere Einzelheiten zur Anleitung zur Probenvorbereitung und zum Service-Workflow wenden Sie sich bitte an aBMKGENE-Experte
Service-Workflow

Experimentdesign

Musterlieferung

Vorexperiment
Räumliche Barcodes

Bibliotheksbau

Sequenzierung

Datenanalyse

Kundendienst
Die von BMKMANU S1000 generierten Daten werden mit der Software „BSTMatrix“ analysiert, die unabhängig von BMKGENE entwickelt wurde, einschließlich:
1) Generierung der Genexpressionsmatrix
2) HE-Bildverarbeitung
3) Kompatibel mit nachgeschalteter Software von Drittanbietern zur Analyse
4) Der Online-„BSTViewer“ hilft dabei, Visualisierungsergebnisse in verschiedenen Auflösungen zu erhalten.
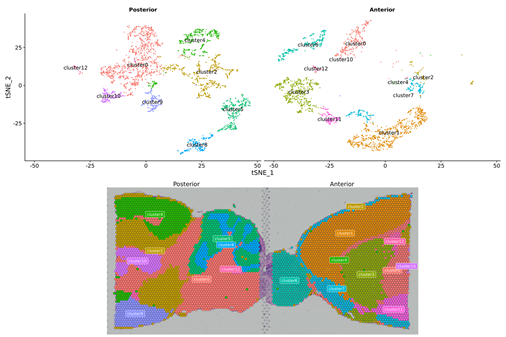
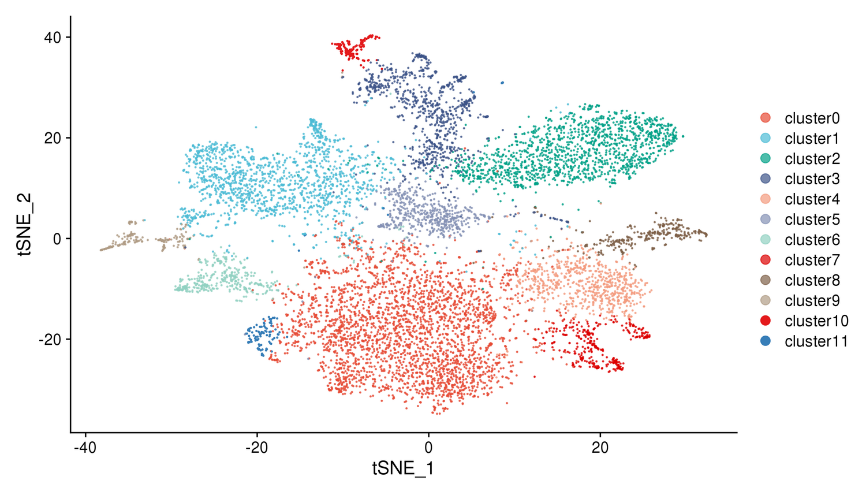
1. Spot-Clustering
2. Räumliche Verteilung
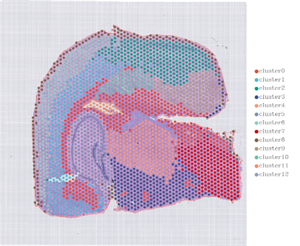
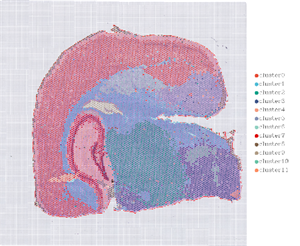
NAnmerkung: AuflösungStufe=13 (100 µm, links); 7 (50 µm, Rechts)
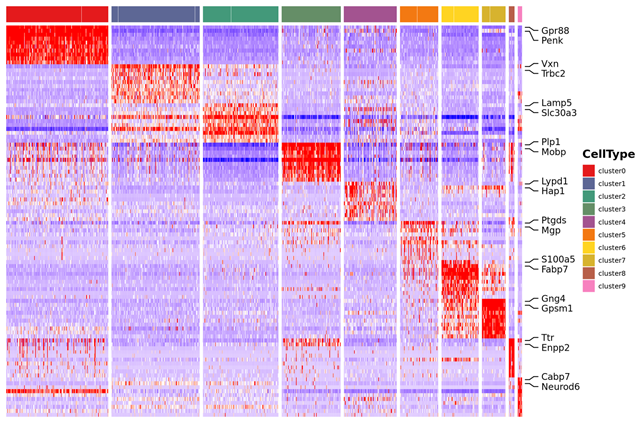
3. Marker-Ausdruckshäufigkeits-Clustering-Heatmap
4. Datenanalyse zwischen Stichproben