Bakterien vervollständigen das Genom
Servicevorteile
● Mehrere Sequenzierungsstrategien für unterschiedliche Forschungsziele verfügbar
● Komplettes Bakteriengenom ohne garantierte Lücken.
● Sehr erfahren in der Zusammenstellung von Bakteriengenomen mit über 10.000 assemblierten vollständigen Genomen.
● Professionelles technisches Kundendienstteam, das spezifischere Forschungsanforderungen erfüllt.
Leistungsbeschreibung
| SequenzierungStrategie | Bibliothek | Qualität garantiert | Geschätzte Bearbeitungszeit |
| Nanopore 100X + Illumina 50X | Nanopore 20K PE150 | 0 Lücke | 30 Tage |
| PacBio HiFi 30X | PacBio 10K |
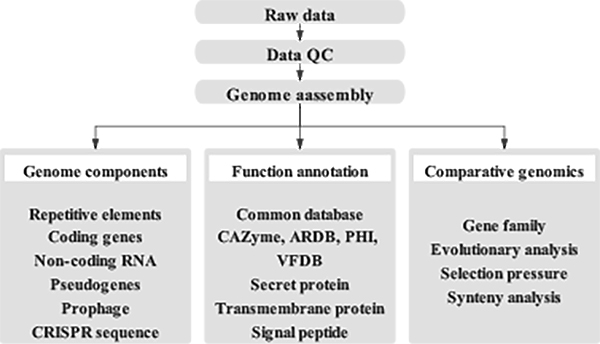
Bioinformatische Analysen
● Qualitätskontrolle der Rohdaten
● Genomassemblierung
● Genomkomponentenanalyse
● Annotation der Genfunktion
● Vergleichende Genomanalyse
Musteranforderungen und Lieferung
Probenanforderungen:
FürDNA-Extrakte:
| Beispielstyp | Menge | Konzentration | Reinheit |
| DNA-Extrakte | > 2 μg | > 20 ng/μl | AD260/280= 1,6-2,5 |
Für Gewebeproben:
| Beispielstyp | Empfohlene Probenbehandlung | Probenlagerung und -versand |
| Bakterien | Beobachten Sie Bakterien unter dem Mikroskop und sammeln Sie Bakterien in ihrer exponentiellen Phase Übertragen Sie die Bakterienkultur (enthält ca. 3–4,5e9 Zellen) in einen 1,5 oder 2 ml Eppendorf.(Auf Eis bleiben) Zentrifugieren Sie das Röhrchen 1 Minute lang bei 14.000 g, um Bakterien zu sammeln, und entfernen Sie den Überstand vorsichtig Verschließen Sie das Röhrchen und frieren Sie die Bakterien mindestens 30 Minuten lang in flüssigem Stickstoff ein.Bewahren Sie das Röhrchen im Kühlschrank bei -80 °C auf. | Frieren Sie die Proben 3-4 Stunden lang in flüssigem Stickstoff ein und lagern Sie sie zur Langzeitkonservierung in flüssigem Stickstoff oder bei -80 Grad.Der Probenversand mit Trockeneis ist erforderlich. |
Service-Workflow

Musterlieferung

Bibliotheksbau

Sequenzierung

Datenanalyse

Kundendienst
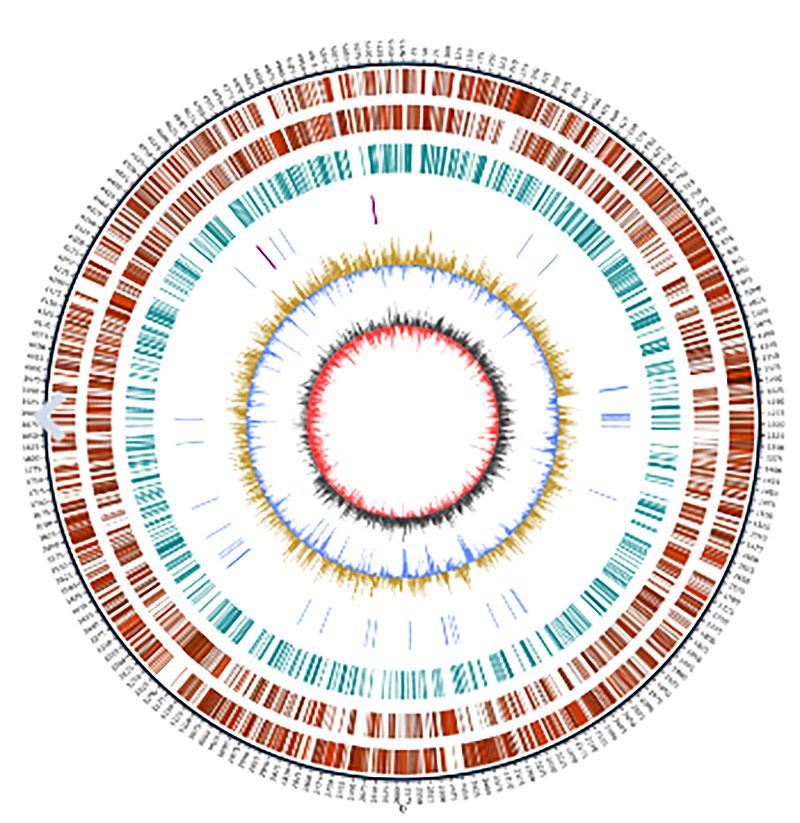
1.Circos des Bakteriengenoms
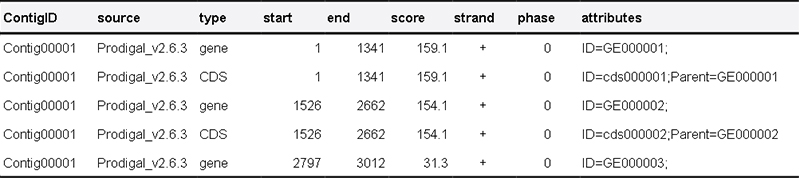
2. Vorhersage kodierender Gene
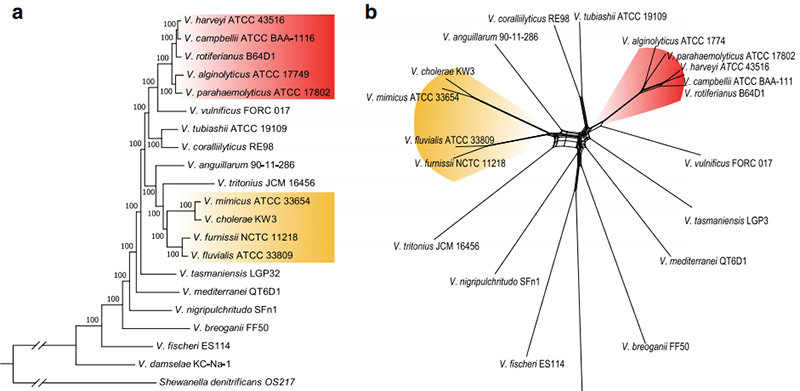
3. Vergleichende Genomanalyse: Stammbaum