
16S/18S/ITS-Amplikonsequenzierung-PacBio
Servicevorteile

● Lange Lesevorgänge, die die Sequenz von 16S/18S/ITS in voller Länge enthüllen
● Hochpräzises Base-Calling mit Sequenzierung im PacBio CCS-Modus
● Auflösung auf Artenebene in der OTU/ASV-Annotation
● Neuester QIIME2-Analysefluss mit vielfältigen Analysen in Bezug auf Datenbank, Annotation, OTU/ASV.
● Anwendbar auf verschiedene mikrobielle Gemeinschaftsstudien
● BMK verfügt über umfassende Erfahrung mit über 100.000 Proben pro Jahr, die Boden, Wasser, Gas, Schlamm, Fäkalien, Därme, Haut, Fermentationsbrühe, Insekten, Pflanzen usw. abdecken.
● BMKCloud erleichterte die Dateninterpretation mit 45 personalisierten Analysetools
Leistungsbeschreibung
| SequenzierungPlattform | Bibliothek | Empfohlene Daten | Seitenwechsel |
| PacBio Fortsetzung II | SMRT-Glocke | 5K/10K/20K-Tags | 44 Arbeitstage |
Bioinformatische Analysen
● Qualitätskontrolle der Rohdaten
● OTU-Clustering/De-Noise (ASV)
● OTU-Anmerkung
● Alpha-Diversität
● Beta-Vielfalt
● Intergruppenanalyse
● Assoziationsanalyse anhand experimenteller Faktoren
● Funktionsgenvorhersage
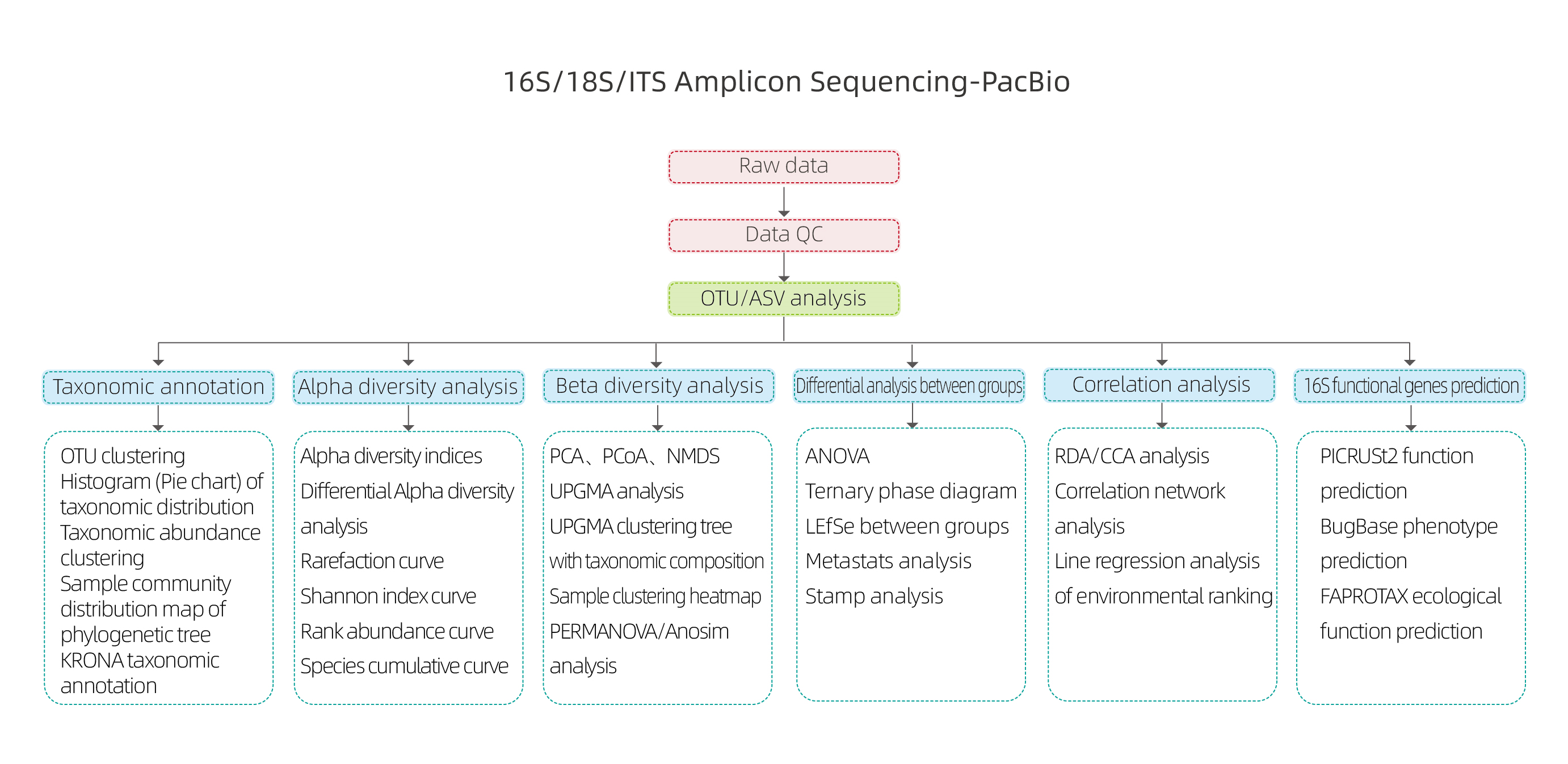
Musteranforderungen und Lieferung
Probenanforderungen:
FürDNA-Extrakte:
| Beispielstyp | Menge | Konzentration | Reinheit |
| DNA-Extrakte | > 1 μg | > 20 ng/μl | AD260/280= 1,6-2,5 |
Für Umweltproben:
| Beispielstyp | Empfohlenes Probenahmeverfahren |
| Boden | Probenmenge: ca.5 g;Verbleibende verwelkte Substanz muss von der Oberfläche entfernt werden;Große Stücke zermahlen und durch einen 2-mm-Filter passieren;Aliquotierte Proben in sterilen EP-Röhrchen oder Zyrotöhrchen zur Reservierung. |
| Kot | Probenmenge: ca.5 g;Sammeln und aliquotieren Sie Proben zur Reservierung in sterilen EP-Röhrchen oder Kryoröhrchen. |
| Darminhalt | Proben müssen unter aseptischen Bedingungen verarbeitet werden.Gesammeltes Gewebe mit PBS waschen;Zentrifugieren Sie das PBS und sammeln Sie den Niederschlag in EP-Röhrchen. |
| Schlamm | Probenmenge: ca.5 g;Sammeln und aliquotieren Sie die Schlammprobe in einem sterilen EP-Röhrchen oder Kryoröhrchen zur Reservierung |
| Gewässer | Für Proben mit einer begrenzten Menge an Mikroben, wie z. B. Leitungswasser, Brunnenwasser usw., sammeln Sie mindestens 1 l Wasser und lassen Sie es durch einen 0,22-μm-Filter laufen, um die Mikroben auf der Membran anzureichern.Bewahren Sie die Membran in einem sterilen Röhrchen auf. |
| Haut | Kratzen Sie die Hautoberfläche vorsichtig mit einem sterilen Wattestäbchen oder einer chirurgischen Klinge ab und legen Sie sie in ein steriles Röhrchen. |
Empfohlene Musterlieferung
Frieren Sie die Proben 3-4 Stunden lang in flüssigem Stickstoff ein und lagern Sie sie zur Langzeitkonservierung in flüssigem Stickstoff oder bei -80 Grad.Der Probenversand mit Trockeneis ist erforderlich.
Service-Workflow

Musterlieferung

Bibliotheksbau

Sequenzierung

Datenanalyse

Kundendienst
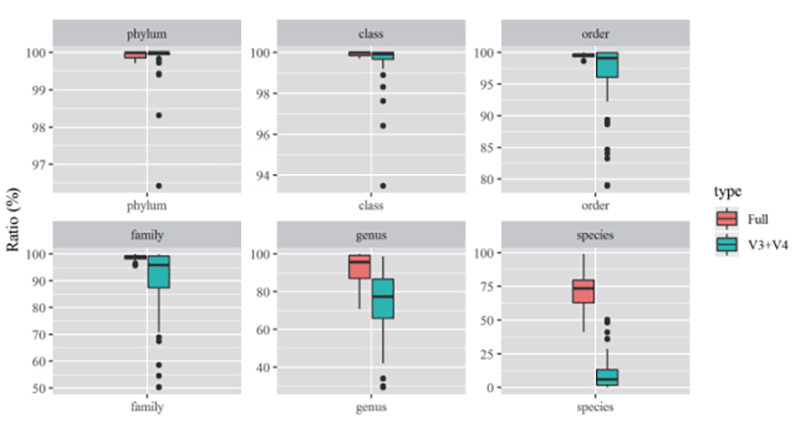
1.Annotationsrate der V3+V4(Illumina)-basierten mikrobiellen Community-Profilierung im Vergleich zur vollständigen (PacBio)-basierten Profilierung.
(Daten von 30 zufällig ausgewählten Projekten wurden für die Statistik herangezogen)
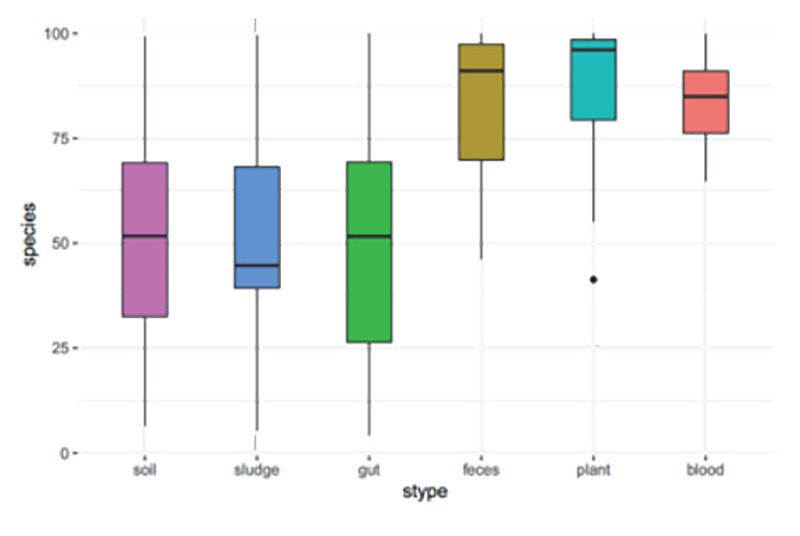
2.Annotationsrate der Amplikonsequenzierung in voller Länge auf Speziesebene in verschiedenen Probentypen
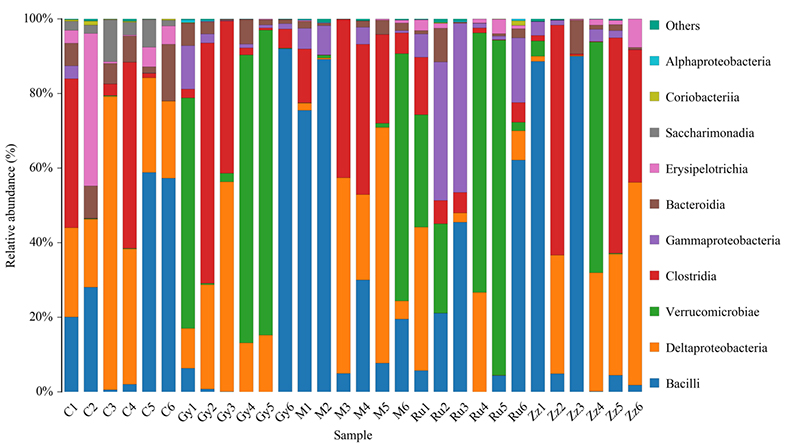
3. Artenverteilung
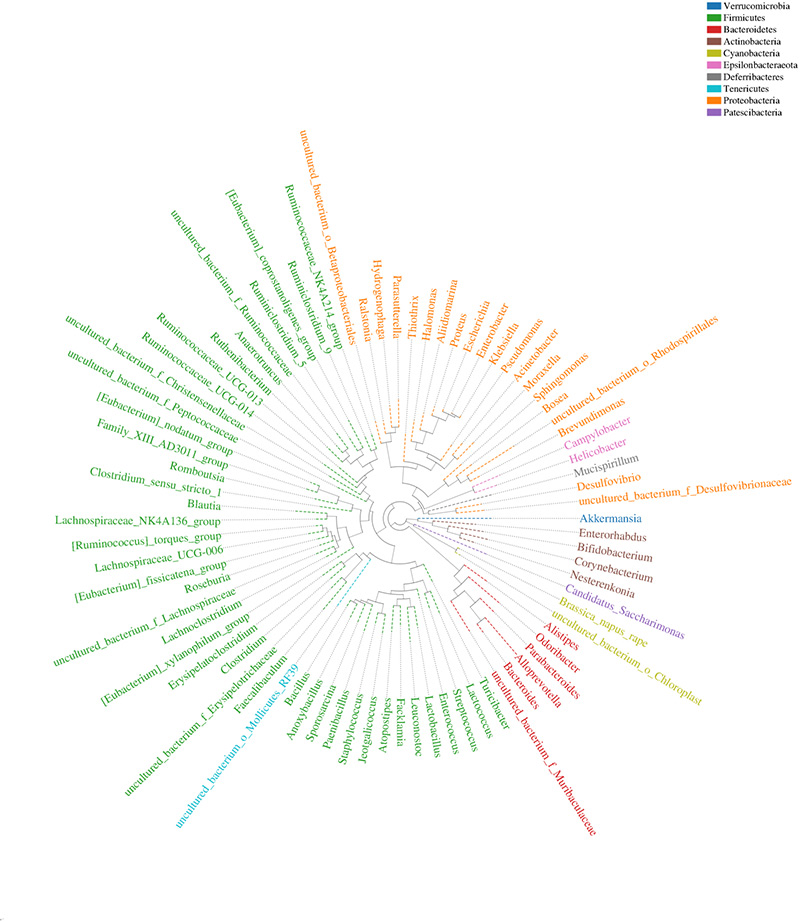
4. Stammbaum
BMK-Fall
Die Exposition gegenüber Arsen führt zu einer Schädigung der Darmbarriere und einer daraus resultierenden Aktivierung der Darm-Leber-Achse, was bei Enten zu Entzündungen und Pyroptose der Leber führt
Veröffentlicht:Wissenschaft der gesamten Umwelt,2021
Sequenzierungsstrategie:
Proben: Kontrollgruppe vs. 8 mg/kg ATO-exponierte Gruppe
Sequenzierungsdatenausbeute: insgesamt 102.583 rohe CCS-Sequenzen
Kontrolle: 54.518 ± 747 effektive CCS
ATO-exponiert: 45.050 ± 1675 effektives CCS
Wichtigste Ergebnisse
Alpha-Diversität:Die ATO-Exposition veränderte den mikrobiellen Reichtum und die Vielfalt im Darm der Enten erheblich.
Metastatanalyse:
Auf Phylum-Ebene: 2 Bakterienstämme nur in Kontrollgruppen nachgewiesen
Auf Gattungsebene: 6 Gattungen unterschieden sich deutlich in der relativen Häufigkeit
Auf Artenebene wurden insgesamt 36 Arten identifiziert, von denen sich 6 in der relaviven Häufigkeit deutlich unterscheiden
Referenz
Thingholm, LB, et al.„Übergewichtige Personen mit und ohne Typ-2-Diabetes zeigen unterschiedliche mikrobielle Funktionsfähigkeit und Zusammensetzung im Darm.“Zellwirt und Mikrobe26.2 (2019).











