GENOMEVOLUTION
naturgenetik
En genomsamling af høj kvalitet fremhæver ruggenomiske egenskaber og agronomisk vigtige gener
PacBio |Illumina |Bionano optisk kort |Hi-C Genome Assembly |Genetisk kort |Selektive fejninger |RNA-Seq |ISO-seq |SLAF-seq
Biomarker Technologies ydede teknisk support til Pacbio-sekventering, Hi-C-sekventering og dataanalyse i denne undersøgelse.
Højdepunkter
1. Det første ruggenom af høj kvalitet på kromosomniveau blev opnået, som har en enkelt kromosomstørrelse større end 1 Gb.
2. Sammenlignet med Tu-, Aet- og Hv-genomet blev en unik nylig LTR-RT-begivenhed observeret i ruggenomet, som var ansvarlig for udvidelsen af ruggenomets størrelse.
3.Divergensen mellem rug og diploid hvede fandt sted efter adskillelsen af byg fra hvede, hvor divergenstiden for de to hændelser var ca. 9,6 og 15 MYA.
Fosforylering af FT-gener kan kontrollere det tidlige overskriftstræk i rug.
4.Selektiv sweep-analyse indikerer mulig involvering af ScID1 i reguleringen af overskriftsdatoen og dens sandsynlige udvælgelse ved domesticering i rug
Baggrund
Baggrund
Rug er en værdifuld føde- og foderafgrøde, en vigtig genetisk ressource til forbedring af hvede og triticale og et uundværligt materiale til effektive komparative genomiske undersøgelser i græsser.Weining rug, en tidlig blomstrende sort dyrket i Kina, er enestående på grund af dens bredspektrede modstandsdygtighed over for både meldug og striberust.For at forstå det genetiske og molekylære grundlag for rugelitegenskaber og for at fremme genomiske undersøgelser og avlsundersøgelser i rug og beslægtede afgrøder, har vi her sekventeret og analyseret genomet af Weining rug.
Præstationer
Rye Genom
Rye-genomet blev konstrueret ved at kæmme PacBio SMRT-aflæsninger, kortlæst Illumina-sekventering såvel som dem fra kromatinkonformationsindfangning (Hi-C), genetisk kortlægning og BioNano-analyse.De samlede contigs (7,74 Gb) tegnede sig for 98,47% af den estimerede genomstørrelse (7,86 Gb), med 93,67% af contigs (7,25 Gb) tildelt syv kromosomer.Gentagne elementer udgjorde 90,31% af det samlede genom.
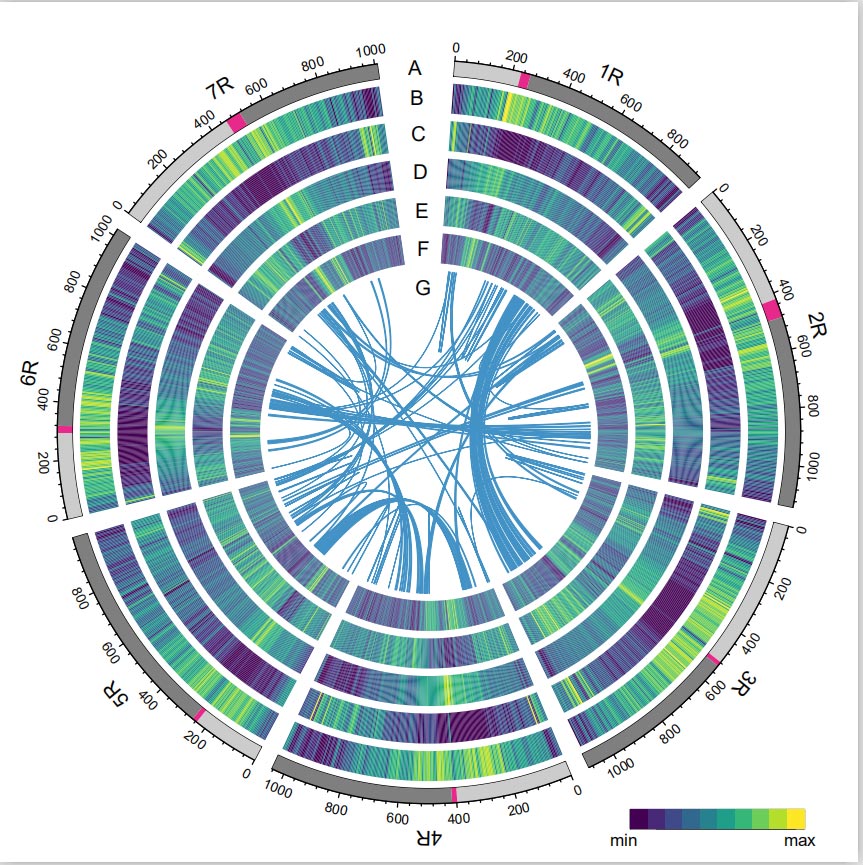
Rye Genom
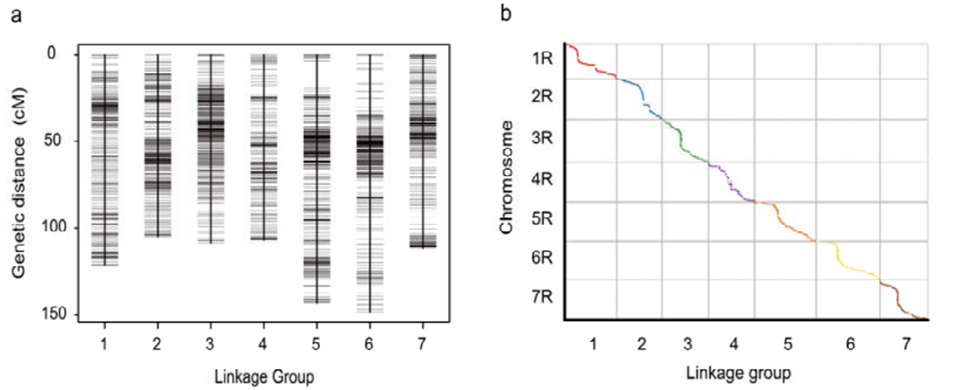
Genetisk koblingskort (WJ) udviklet ved hjælp af 295 F2-planter afledt af krydsning af to ruglandracer (Weining × Jingzhou)
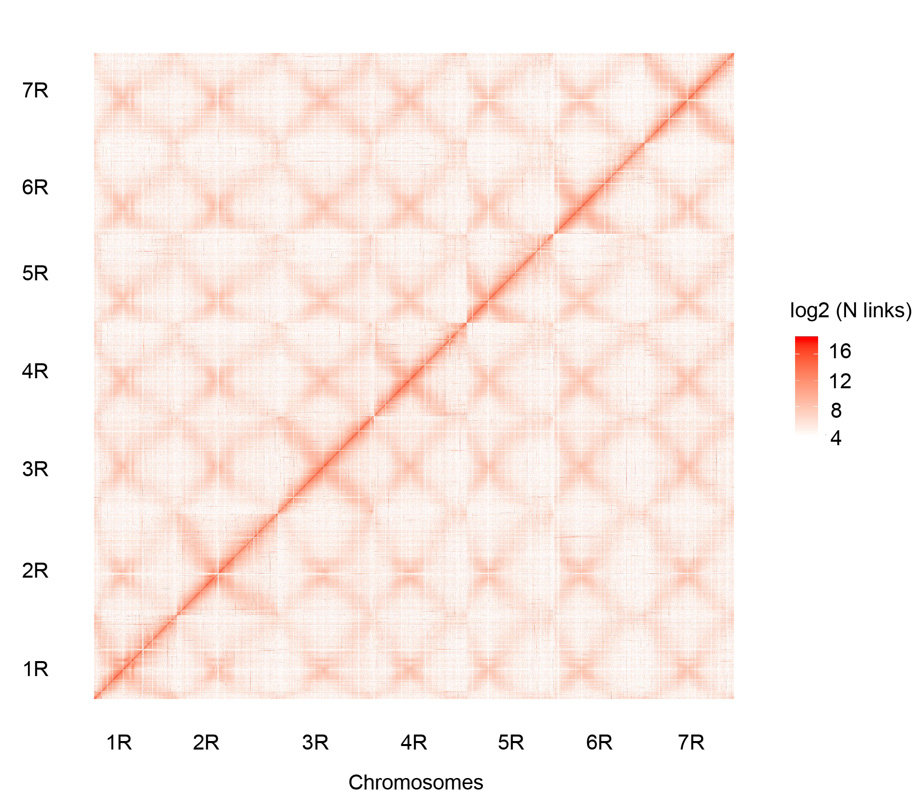
Hi-C kontaktkort over de syv samlede Weining rugkromosomer (1R – 7R)
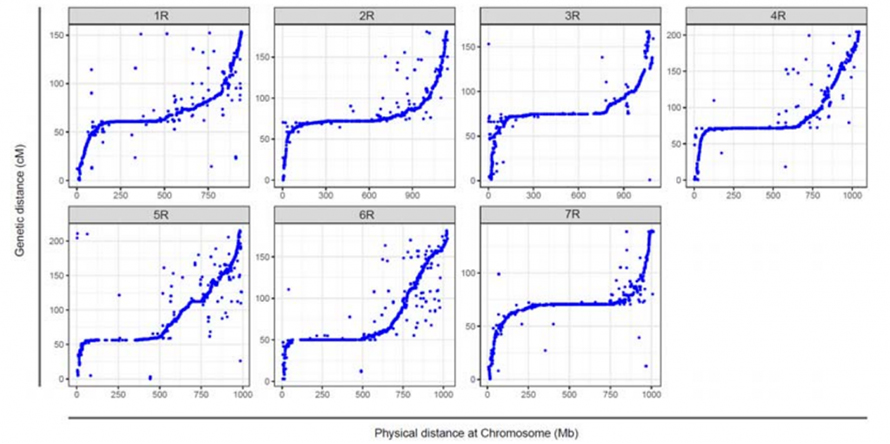
Justering mellem de syv samlede kromosomer af Weining rug og de syv rugbindingsgrupper udviklet ved hjælp af Lo7 x Lo255 RIL-population
LTR Assembly Index (LAI) værdien af ruggenomet blev fundet at være 18,42, og 1.393 (96.74%) af de 1.440 meget konserverede BUSCO gener blev identificeret ud. Disse resultater tyder på, at Weining ruggenomsekvensen er af høj kvalitet i både intergeniske gener. og generiske regioner.I alt 86.991 proteinkodende gener, herunder 45.596 gener med høj konfidens (HC) og 41.395 gener med lav konfidens (LC) blev forudsagt.
2. Analyse af TE'er
Analyse af TE'er.I alt 6,99 Gb, der repræsenterer 90,31% af Weining-forsamlingen, blev annoteret som TE'er, som omfattede 2.671.941 elementer tilhørende 537 familier.Dette TE-indhold var klart højere end det tidligere rapporterede for Ta (84,70%), Tu (81,42%), Aet (84,40%), WEW (82,20%) eller Hv (80,80%).De lange terminale gentagne retrotransposoner (LTR-RT'er), inklusive Gypsy, Copia og uklassificerede RT-elementer, var de dominerende TE'er, og 1 optog 84,49 % af annoteret TE-indhold og 76,29 % af det samlede Weining-genom;CACTA DNA-transposoner var de næstmest udbredte TE'er, der udgjorde 11,68% af annoteret TE-indhold og 10,55% af det samlede Weining-genom.
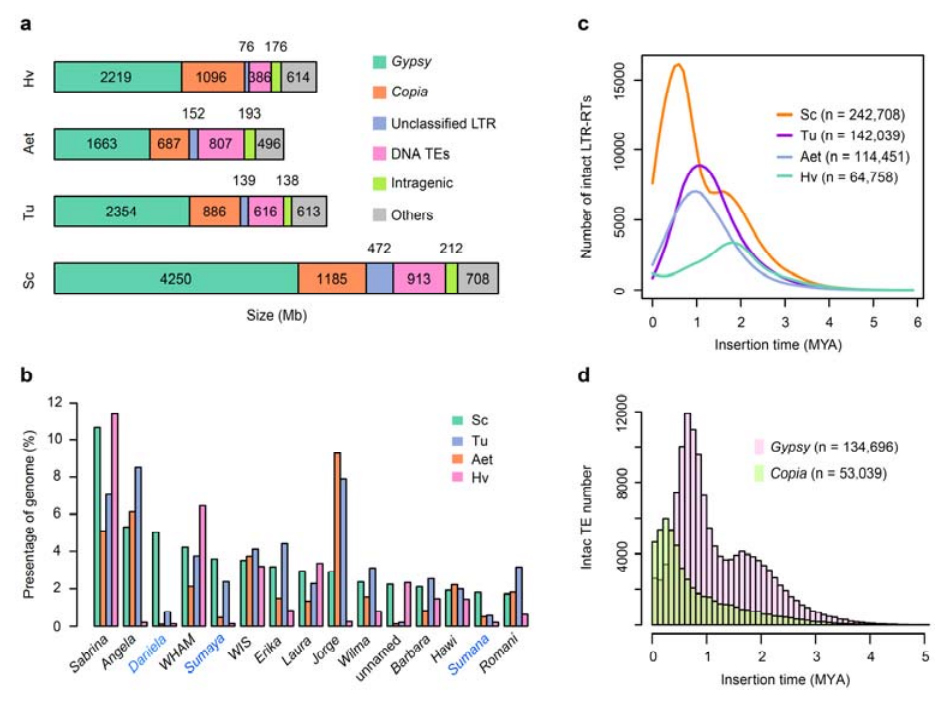
Analyse af transposon elementer af rug
Weining rug havde en forholdsvis høj andel af nylige indsættelser af LTR-RT'er med toppen af amplifikationen for omkring 0,5 millioner år siden (MYA), som var den seneste blandt de fire arter;den anden top, forekom ca. 1,7 MYA, var ældre og også set i byg.På superfamilieniveau blev der fundet meget nylige udbrud af Copia-elementer i Weining-rug ved 0,3 MYA, mens forstærkningerne af Gypsy RT'er dominerende formede det bimodale distributionsmønster af LTR-RT-burstdynamik.
3. Undersøgelse af ruggenomudvikling og kromosomsyntenier
Divergensen mellem rug og diploid hvede fandt sted efter adskillelsen af byg fra hvede, hvor divergenstiden for de to hændelser var henholdsvis ca. 9,6 og 15 MYA.1R, 2R, 3R var helt kollineære med henholdsvis gruppe 1, 2 og 3 kromosomer af hvede.4R, 5R, 6R, 7R blev fundet, at der eksisterer fusioner og segmenter i stor skala.
4. Analyse af genduplikationer og deres indvirkning på stivelsesbiosyntesegener
Navnlig var antallet af tandemduplikerede gener (TDG'er) og proksimalt duplikerede gener (PDG'er) af Weining rug begge højere end dem, der blev fundet for Tu, Aet, Hv, Bd og Os.Transponerede duplikerede gener (TrDG'er) var også flere end dem, der specifikt blev fundet for Tu og Aet.Ruggenomudvidelse er ledsaget af højere antal genduplikationer.De øgede TE-udbrud i rug kan have ført til et forhøjet antal TrDG'er.
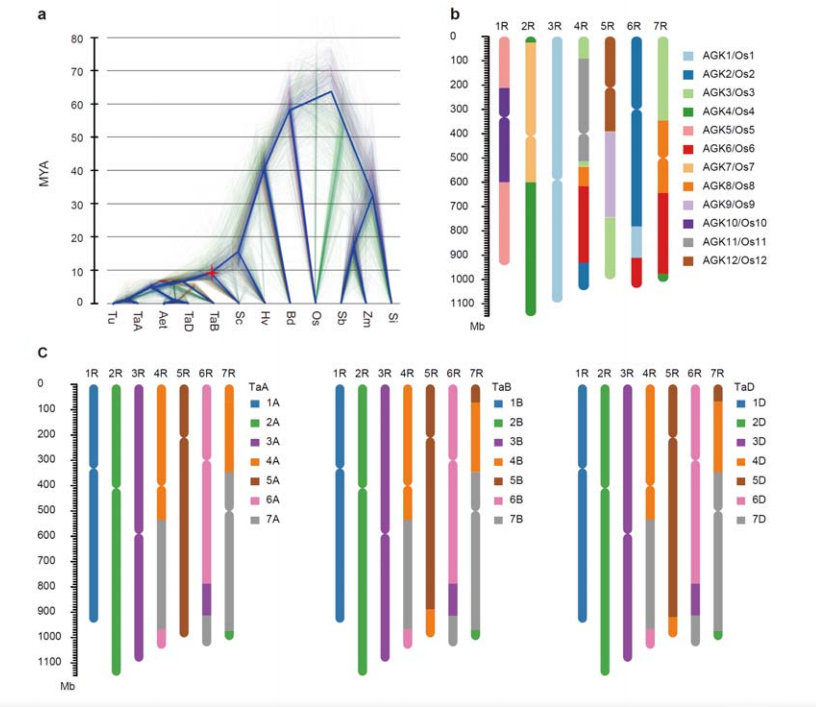
Evolutionære og kromosomsyntetiske analyser af ruggenom
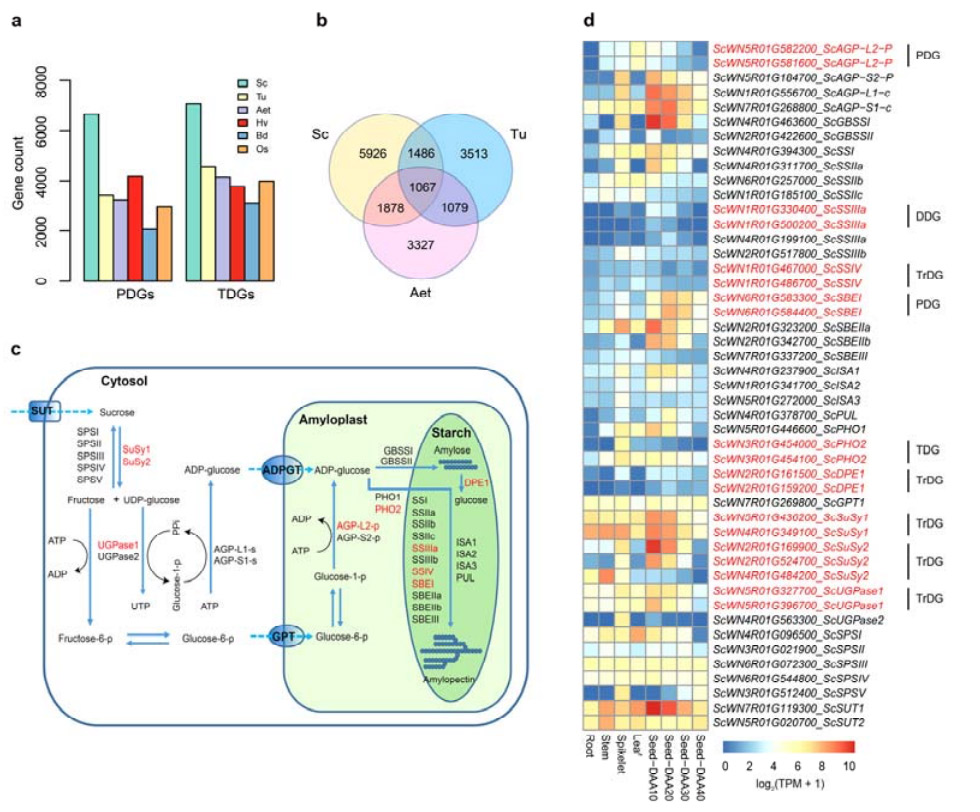
Analyse af ruggenduplikationer og deres indvirkning på mangfoldigheden af stivelsesbiosynteserelaterede gener (SBRG'er)
5. Dissektion af rugfrølagringsprotein (SSP) genloci
Fire kromosomale loci (Sec-1 til Sec-4), der specificerer rug-SSP'er, er blevet identificeret på 1R eller 2R.α-gliadingener blev først udviklet for nylig i hvede og nært beslægtede arter efter divergensen mellem hvede og rug.
6. Undersøgelse af transkriptionsfaktor (TF) og sygdomsresistensgener
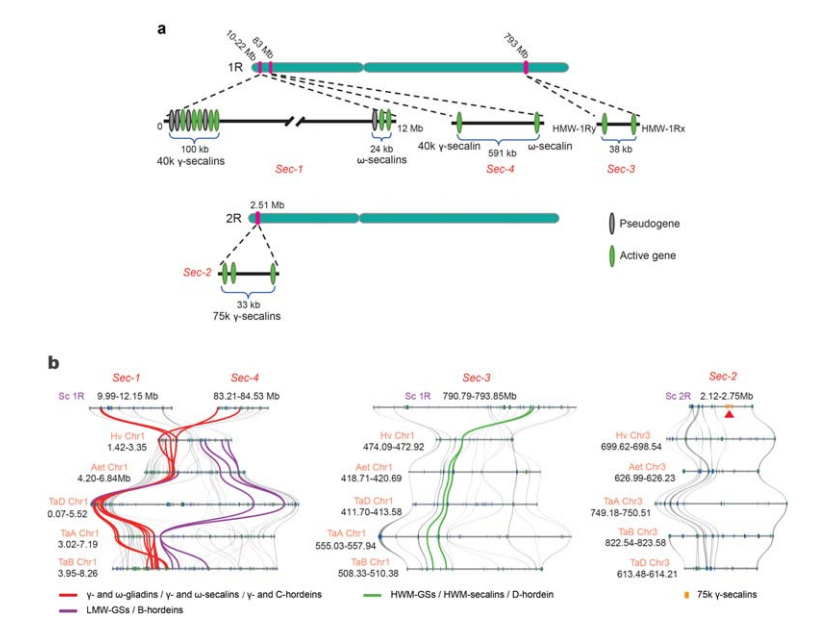
Analyse af rug secalin loci
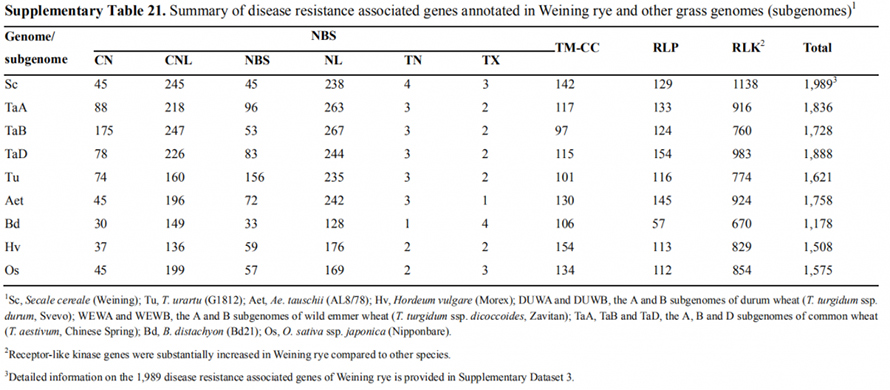
Weingrug havde flere sygdomsresistensassocierede (DRA) gener (1.989, supplerende data 3) end Tu (1.621), Aet (1.758), Hv (1.508), Bd (1.178), Os (1.575) og A (1.836) ), B (1.728) og D (1.888) subgenomer af almindelig hvede.
7. Undersøgelse af genekspressionstræk forbundet med tidlige overskriftsegenskaber
To FT-gener med relativt høj ekspression under langdagsforhold, ScFT1 og ScFT2, blev annoteret i Weining-genomsamling.To aminosyrerester af ScFT2 (S76 og T132) phosphorylering blev fundet sammenhæng med sænkende tidskontrol
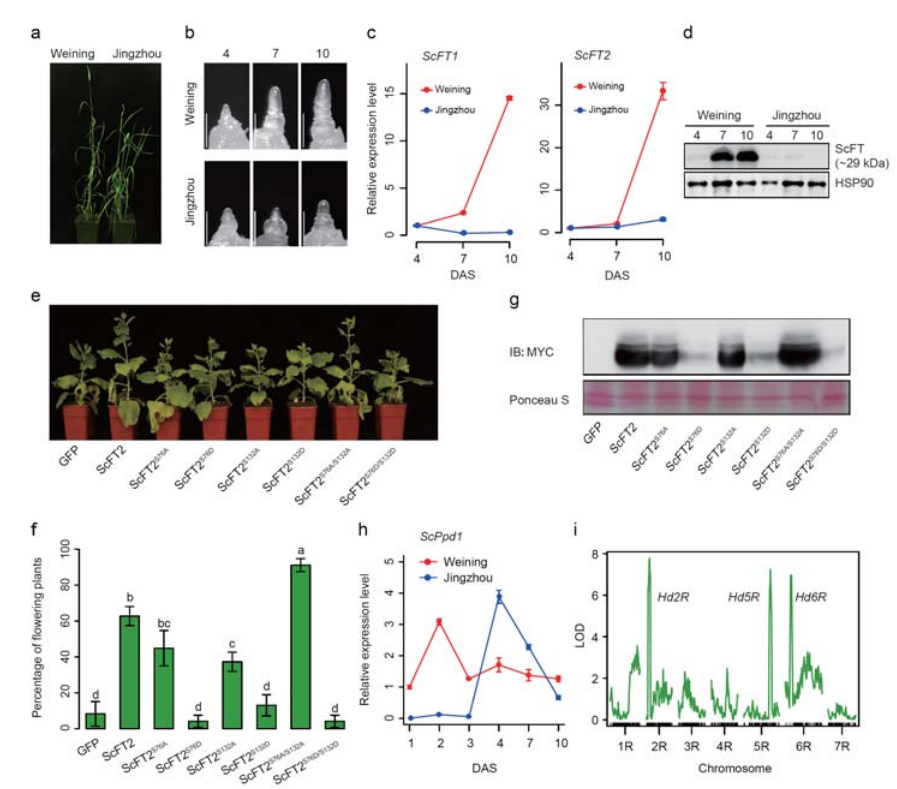
Udviklings- og genekspressionstræk forbundet med den tidlige overskriftsegenskab af Weining rug
8. Udvinding af kromosomale regioner og lokaliteter, der potentielt er involveret i rug-domestisering
I alt 123.647 SNP'er blev brugt til at udføre selevtive sweep-analyse mellem kultiveret rug og S. vavilovii.11 selektive sweep-signaler identificeret ved reduktionsindeks (DRI), fikseringsindeks (FST) og XP-CLR-metode.ScID1 blev fundet mulig involvering i reguleringen af overskriftsdato.
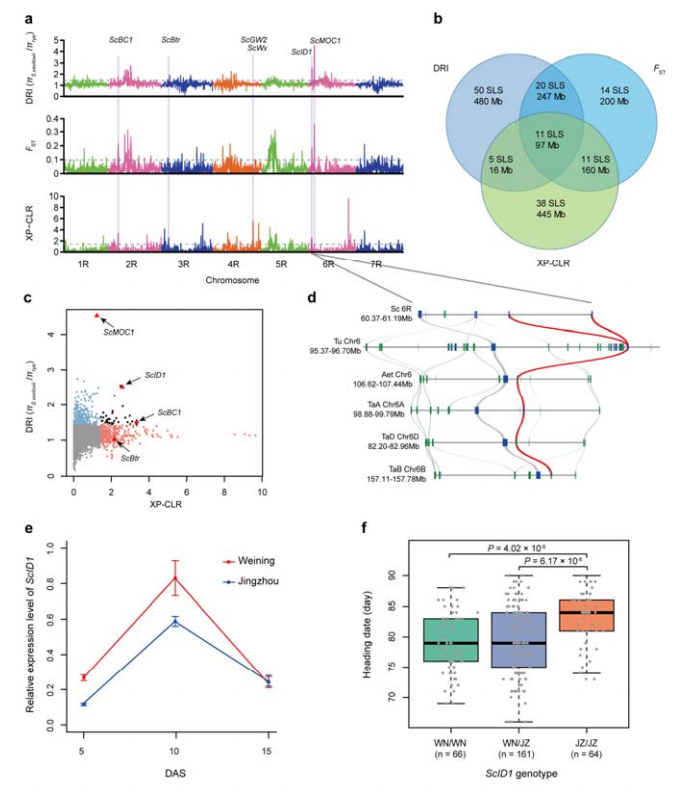
Identifikation og analyse af kromosomale regioner og loci, der potentielt er relateret til rugdomestisering
Reference
Li GW et al.En genomsamling af høj kvalitet fremhæver ruggenomiske egenskaber og agronomisk vigtige gener.Naturgenetik (2021)
Nyheder og højdepunkter sigter på at dele de seneste succesfulde cases med Biomarker Technologies, fange nye videnskabelige resultater såvel som fremtrædende teknikker anvendt under undersøgelsen.
Indlægstid: Jan-05-2022

