Human Whole Exome Sequencing
Service fordele
● Målrettet proteinkodningsregion: ved at indfange og sekventere proteinkodende region, bruges hWES til at afsløre varianter relateret til proteinstruktur;
● Høj nøjagtighed: med høj sekventeringsdybde letter hWES detektion af almindelige varianter og sjældne varianter med frekvenser lavere end 1 %;
● Omkostningseffektiv: hWES giver ca. 85 % af humane sygdomsmutationer fra 1 % af det menneskelige genom;
● Fem strenge QC-procedurer, der dækker hele processen med Q30>85% garanteret.
Eksempel specifikationer
| Platform
| Bibliotek
| Exon Capture Strategy
| Anbefal sekvenseringsstrategi
|
|
Illumina NovaSeq platform
| PE150 | Agilent SureSelect Human All Exon V6 IDT xGen Exome Hyb Panel V2 | 5 Gb 10 Gb |
Prøvekrav
| Prøvetype
| Beløb(Qubit®)
| Bind
| Koncentration
| Renhed (NanoDrop™) |
|
Genomisk DNA
| ≥ 300 ng | ≥ 15 μL | ≥ 20 ng/μL | OD260/280=1,8-2,0 ingen nedbrydning, ingen forurening
|
Anbefalet sekventeringsdybde
For Mendelske lidelser/sjældne sygdomme: effektiv sekventeringsdybde over 50×
For tumorprøver: effektiv sekventeringsdybde over 100×
Bioinformatik


Service Work Flow

Prøve levering

DNA-ekstraktion

Biblioteksbyggeri

Sekvensering

Dataanalyse

Data levering

Eftersalgsservice
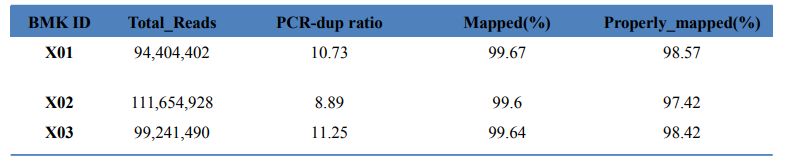
1.Opstillingsstatistikker
Tabel 1 Statistik over kortresultat
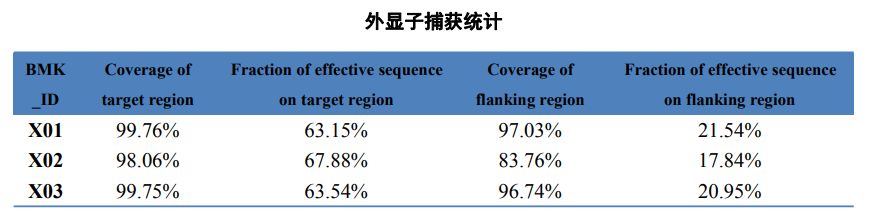
Tabel 2 Statistik over exome capture
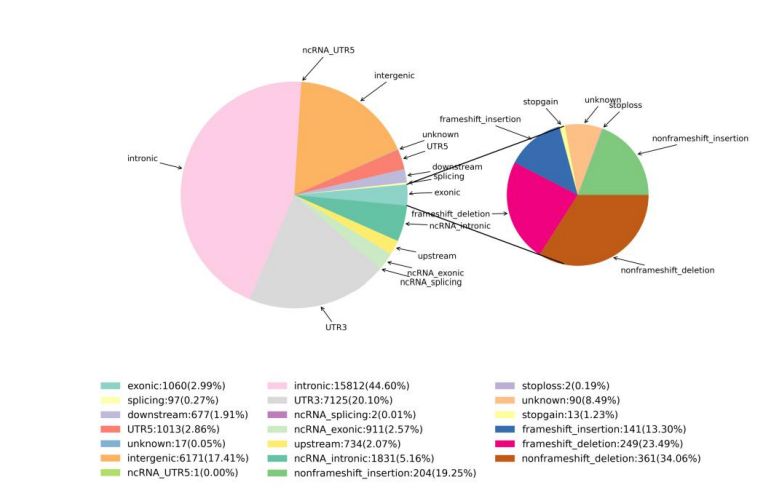
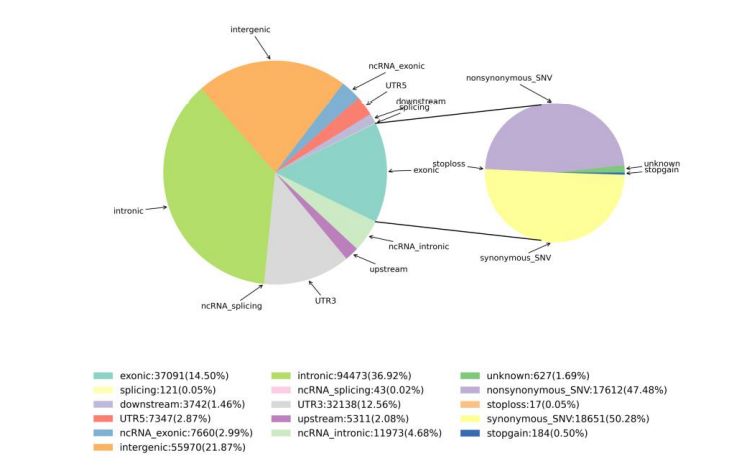
2. Variationsdetektion
Figur 1 Statistik for SNV og InDel
3.Avanceret analyse
Figur 2 Circos-plot af genom-dækkende skadelig SNV og InDel
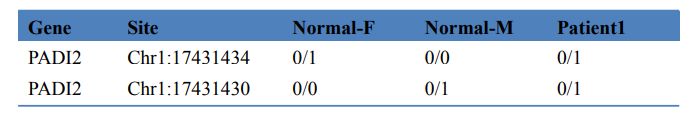
Tabel 3 Screening af sygdomsfremkaldende gener