Sequenza di l'exoma tutale umanu
Vantaghji di serviziu
● Targeted Protein Coding Region: da catturà è sequencing regione codifica proteina, hWES hè utilizatu per revelà varianti ligati à a struttura di a proteina;
● High Accuracy: cù alta prufundità di sequenza, hWES facilita a deteczione di varianti cumuni è varianti rari cù frequenze più bassu di 1%;
● Cost Effective: hWES rende circa 85% di mutazioni di malatie umane da 1% di u genoma umanu;
● Cinque prucedure QC strette chì copre tuttu u prucessu cù Q30> 85% guarantisci.
Specificazioni di mostra
| Piattaforma
| Biblioteca
| Strategia di cattura di Exon
| Cunsigliu Strategia Sequencing
|
|
Piattaforma Illumina NovaSeq
| PE150 | Agilent SureSelect Human All Exon V6 IDT xGen Exome Hyb Panel V2 | 5 Gb 10 Gb |
Requisiti di mostra
| Tipu di mostra
| A quantità(Qubit®)
| Volume
| Cuncentrazione
| Purezza (NanoDrop™) |
|
DNA genomicu
| ≥ 300 ng | ≥ 15 μL | ≥ 20 ng/μL | OD260/280 = 1,8-2,0 senza degradazione, nè contaminazione
|
Profundità di sequenza cunsigliata
Per i disordini mendeliani / malatie rari: prufundità di sequenza efficace sopra 50 ×
Per campioni di tumore: prufundità di sequenza efficace sopra 100 ×
Bioinformatica


Flussu di travagliu di serviziu

Consegna di mostra

estrazione di DNA

Custruzzione di biblioteca

Sequencing

Analisi di dati

Consegna di dati

Servizi post-vendita
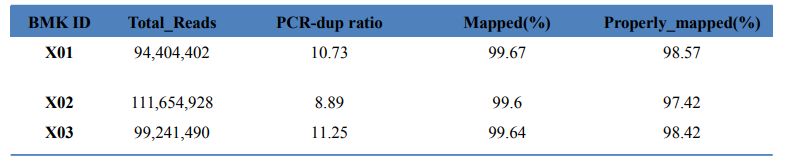
1.Alignment statistiche
Table 1 Statistics of map result
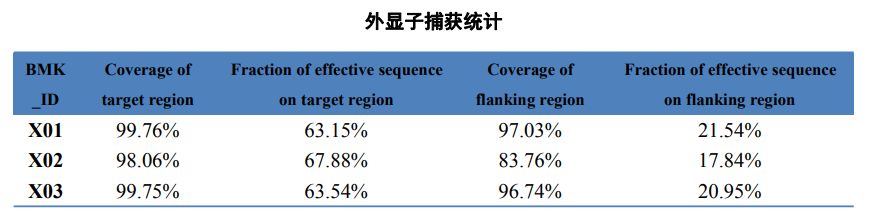
Tabella 2 Statistiche di cattura di l'esoma
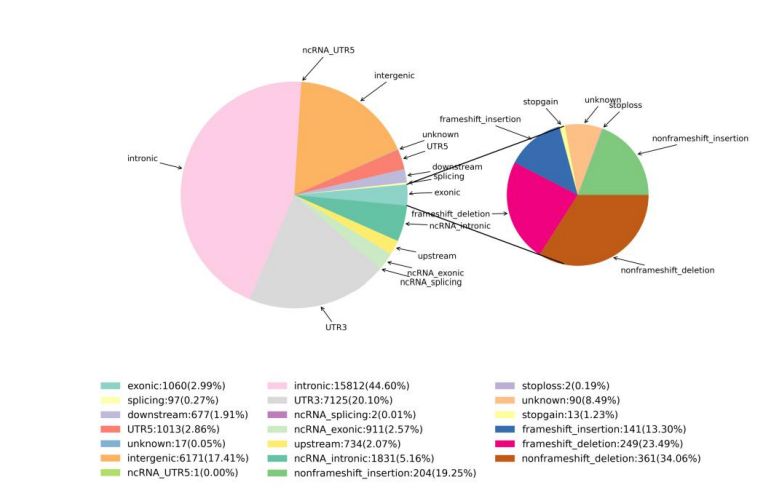
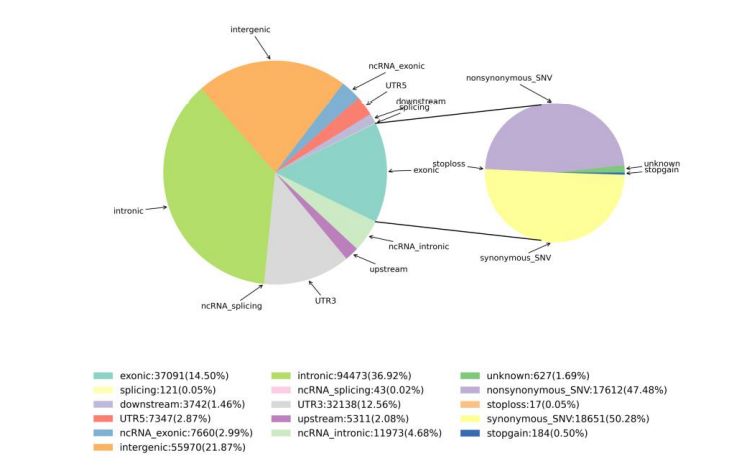
2.Variation Detection
Figura 1 Statistiche di SNV è InDel
3.Analisi avanzata
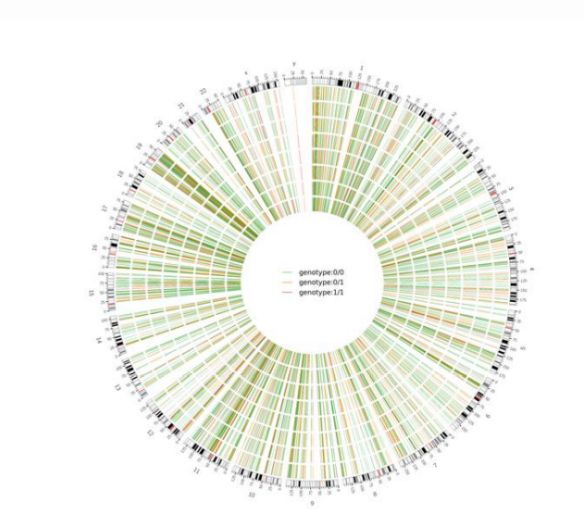
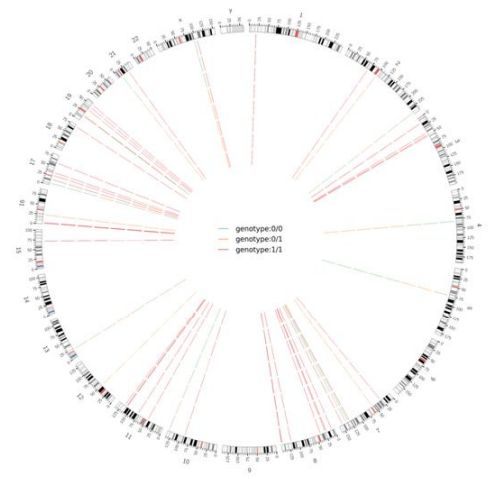
Figura 2 Circos trama di Genome-wide dannusu SNV è InDel
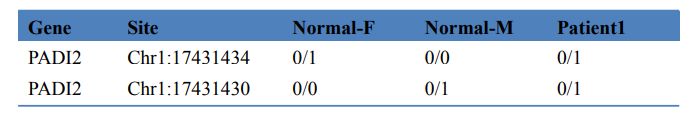
Table 3 Screening di i geni chì causanu a malatia