
Pagsunodsunod sa Genome sa Tanum/Animal De Novo
Mga Bentaha sa Serbisyo

Pag-uswag sa mga sequencing platform ug bioinformatics sade novoasembliya sa genome
(Amarasinghe SL et al.,Genome Biology, 2020)
● Pagtukod ug bag-ong mga genome ug pagpaayo sa kasamtangan nga reference genome alang sa mga matang sa interes.
● Mas taas nga katukma, pagpadayon ug pagkakompleto sa asembliya
● Pagtukod ug sukaranan nga kahinguhaan para sa panukiduki sa sequence polymorphism, QTLs, gene editing, breeding, etc.
● Gisangkapan sa bug-os nga spectrum sa ikatulo nga henerasyon nga sequencing platform: one-stop genome assembly solution
● Flexible sequencing ug assembling nga mga estratehiya sa pagtuman sa lain-laing mga genome uban sa lain-laing mga bahin
● Taas nga hanas nga bioinformatician team nga adunay daghang kasinatian sa komplikado nga genome assemblies, lakip ang polyploid, higante nga genome, ug uban pa.
● Kapin sa 100 ka malampuson nga mga kaso nga adunay accumulative published impact factor nga kapin sa 900
● Ang turn-around-time nga ingon ka paspas sa 3 ka bulan alang sa chromosome-level genome assembly.
● Solid nga teknikal nga suporta nga adunay sunod-sunod nga mga patente ug software copyrights sa duha ka eksperimental nga bahin ug bioinformatics.
Mga Detalye sa Serbisyo
|
Kontento
|
Plataporma
|
Gidugayon sa pagbasa
|
Coverage
|
| Genome Survey
| Illumina NovaSeq
| PE150
| ≥ 50X
|
| Genome Sequencing
| PacBio Revio
| 15 kb HiFi Reads
| ≥ 30X
|
| Hi-C
| Illumina NovaSeq
| PE150
| ≥100X
|
Daloy sa trabaho

Sample nga Kinahanglanon ug Paghatud
Sample nga Kinahanglanon:
| Mga espisye | Tissue | Para sa PacBio | Alang sa Nanopore |
| Mga mananap | Mga organo sa visceral (atay, spleen, ug uban pa) | ≥ 1.0 g | ≥ 3.5 g |
| Kaunuran | ≥ 1.5 g | ≥ 5.0 g | |
| Dugo sa mga mammal | ≥ 1.5 mL | ≥ 5.0 mL | |
| Dugo sa isda o langgam | ≥ 0.2 mL | ≥ 0.5 mL | |
| Mga tanom | Lab-as nga mga dahon | ≥ 1.5 g | ≥ 5.0 g |
| Petal o tukog | ≥ 3.5 g | ≥ 10.0 g | |
| Mga gamot o liso | ≥ 7.0 g | ≥ 20.0 g | |
| Mga selula | Kultura sa selula | ≥ 3×107 | ≥ 1×108 |
Girekomenda nga Sample Delivery
Kontainer: 2 ml centrifuge tube (Tin foil dili girekomendar)
Alang sa kadaghanan sa mga sample, among girekomenda nga dili tipigan sa ethanol.
Sample nga pag-label: Ang mga sample kinahanglan nga klaro nga gimarkahan ug parehas sa gisumite nga porma sa impormasyon sa sample.
Pagpadala: Dry-ice: Ang mga sample kinahanglan nga ibutang una sa mga bag ug ilubong sa dry-ice.
Daloy sa Trabaho sa Serbisyo

Eksperimento nga disenyo

Sample nga paghatod

Pagkuha sa DNA

Pagtukod sa librarya

Pagsunodsunod

Pagtuki sa datos

After-sale nga serbisyo
* Ang mga resulta sa demo nga gipakita dinhi tanan gikan sa mga genome nga gipatik sa Biomarker Technologies
1.Circos sa chromosome-level genome assembly saG. rotundifoliumpinaagi sa Nanopore sequencing platform
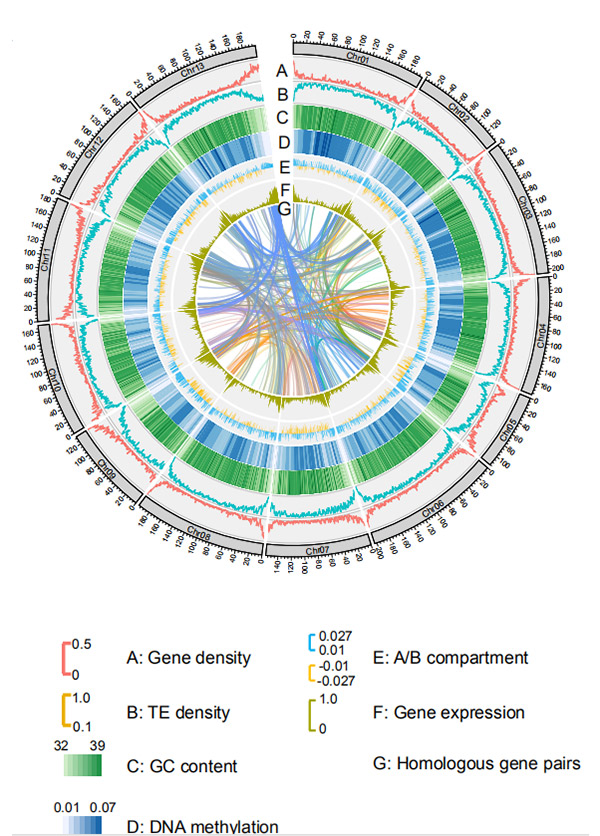
Wang M et al.,Molecular Biology ug Evolution, 2021
2.Estadistika sa Weining rye genome assembly ug anotasyon
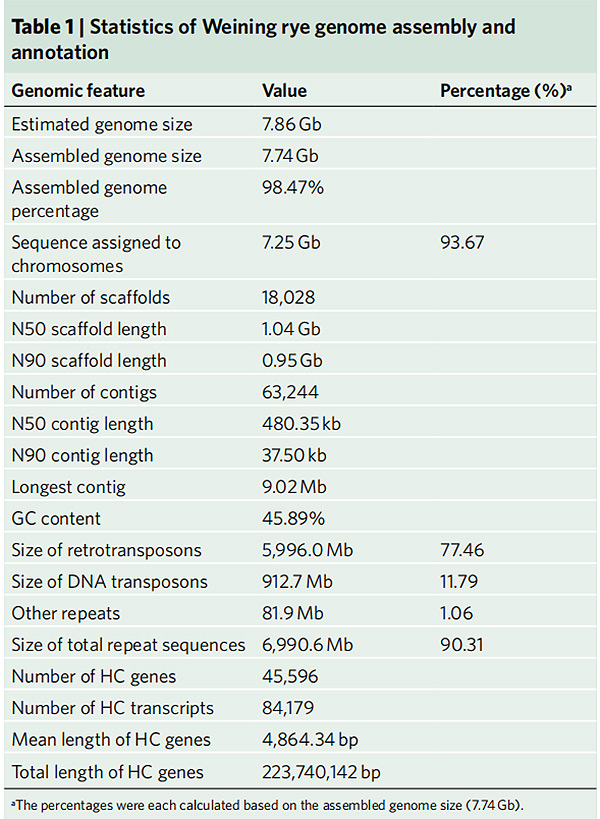
Li G et al.,Mga Genetika sa Kinaiyahan, 2021
3.Pagtagna sa gene saSechium edulegenome, nakuha gikan sa tulo ka mga pamaagi sa pagtagna:De novopanagna, Homology-based nga prediksyon ug RNA-Seq data base prediksiyon
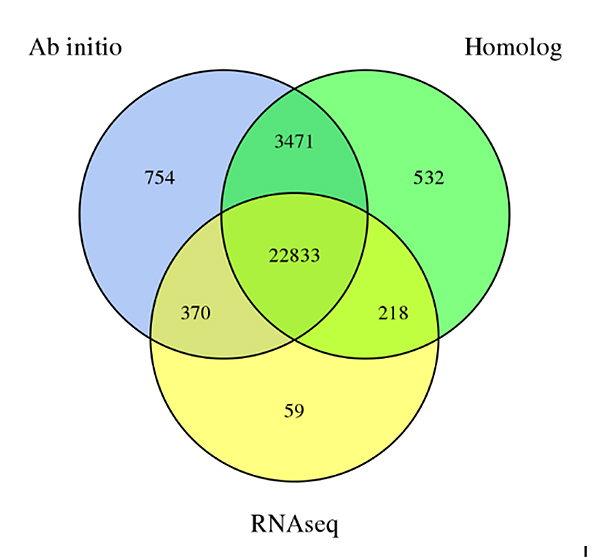
Fu A et al.,Pagpanukiduki sa Hortikultura, 2021
4. Pag-ila sa intact long terminal repeats sa tulo ka cotton genome
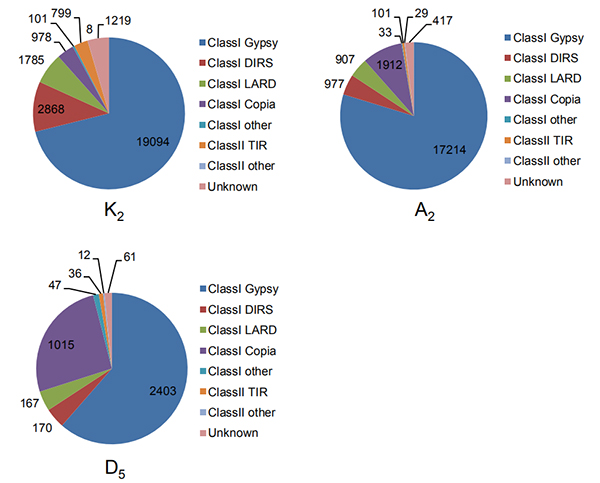
Wang M et al.,Molecular Biology ug Evolution, 2021
5.Hi-C init nga mapa saC. acuminatagenome nga nagpakita sa genome-wide all-by-all interaction.Ang intensity sa mga interaksyon sa Hi-C kay proporsyonal sa linear nga gilay-on tali sa contigs.Ang usa ka limpyo nga tul-id nga linya sa kini nga mapa sa init nagpakita sa usa ka tukma kaayo nga pag-angkla sa mga contigs sa mga chromosome.(Contig anchoring ratio: 96.03%)
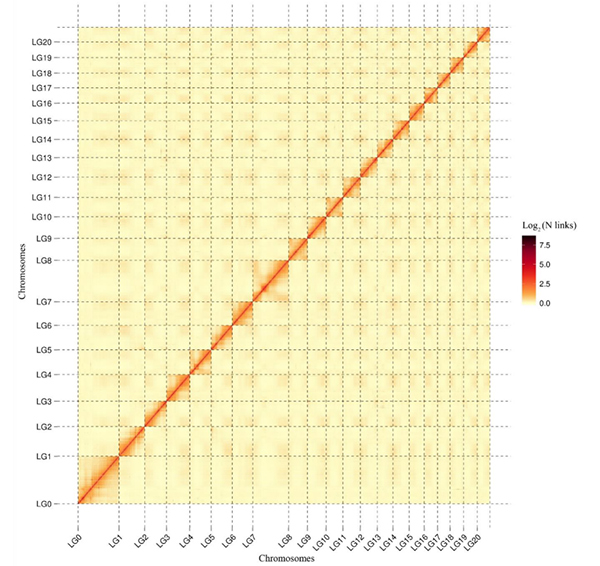
kang M et al.,Komunikasyon sa Kinaiyahan,2021
Kaso sa BMK
Ang usa ka taas nga kalidad nga genome assembly nagpasiugda sa rye genomic nga mga kinaiya ug agronomically importante nga mga gene
Gipatik: Mga Genetika sa Kinaiyahan, 2021
Estratehiya sa pagkasunodsunod:
Genome assembly: PacBio CLR mode nga adunay 20 kb library (497 Gb, gibanabana nga 63×)
Pagtul-id sa han-ay: NGS nga adunay 270 bp DNA library (430 Gb, gibanabana nga 54 ×) sa plataporma sa Illumina
Pag-angkla sa Contigs: Hi-C library(560 Gb, gibanabana nga 71×) sa plataporma sa Illumina
Optical nga mapa: (779.55 Gb, gibanabana nga 99 ×) sa Bionano Irys
Pangunang mga resulta
1. Usa ka asembliya sa Weining rye genome ang gipatik nga adunay kinatibuk-ang gidak-on sa genome nga 7.74 Gb(98.74% sa gibanabana nga gidak-on sa genome pinaagi sa flow cytometry).Ang Scaffold N50 niini nga asembliya nakab-ot ang 1.04 Gb.93.67% sa contigs malampuson nga nakaangkla sa 7 pseudo-chromosome.Kini nga asembliya gisusi pinaagi sa linkage map, LAI ug BUSCO, nga miresulta sa taas nga mga marka sa tanan nga mga ebalwasyon.
2. Ang dugang nga mga pagtuon sa comparative genomics, genetic linkage map, transcriptomics nga mga pagtuon gihimo base niini nga genome.Usa ka serye sa mga kinaiya nga may kalabutan sa genomic nga mga bahin ang gipadayag lakip ang genome-wide gene duplications ug ang ilang epekto sa starch biosynthesis genes;Pisikal nga organisasyon sa komplikadong prolamin loci, gene nga ekspresyon nagpakita sa nagpahipi nga sayo nga ulohan nga kinaiya ug putative domestication-kauban chromosomal rehiyon ug loci sa rye.
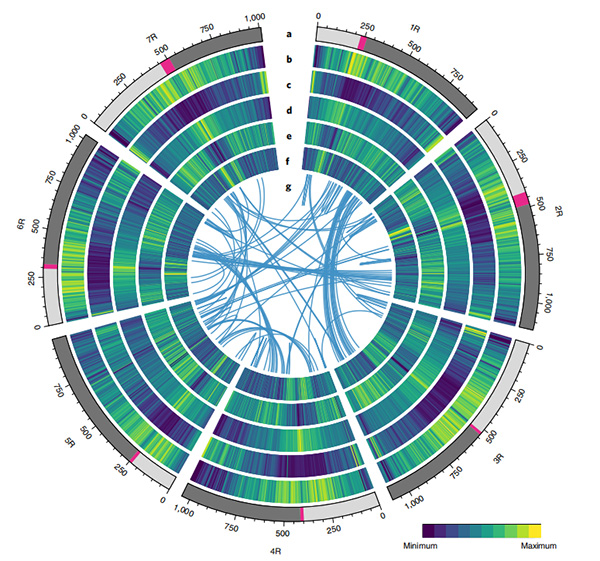 Circos diagram sa genomic features sa Weining rye genome | 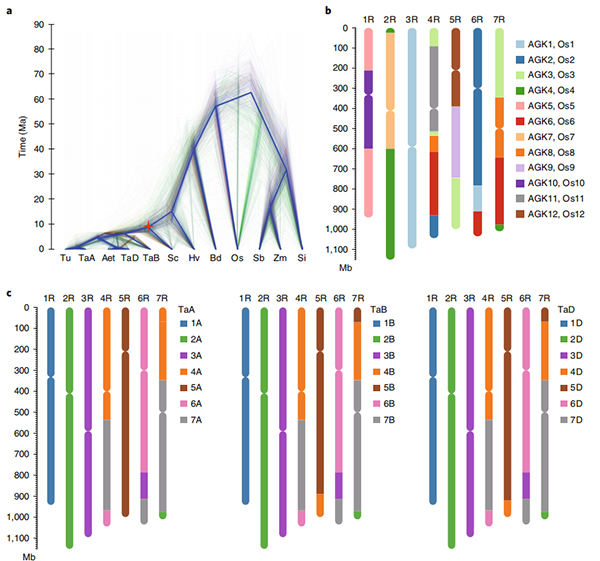 Pag-analisar sa ebolusyonaryo ug chromosome synteny sa rye genome |
Li, G., Wang, L., Yang, J.ug uban pa.Ang usa ka taas nga kalidad nga genome assembly nagpasiugda sa rye genomic nga mga kinaiya ug agronomically importante nga mga gene.Nat Genet 53,574–584 (2021).
https://doi.org/10.1038/s41588-021-00808-z










