GENOME EVOLUTION
genetics sa kinaiyahan
Ang usa ka taas nga kalidad nga genome assembly nagpasiugda sa rye genomic nga mga kinaiya ug agronomically importante nga mga gene
PacBio |Illumina |Bionano Optical Map |Hi-C Genome Assembly |Genetikong Mapa |Pinili nga mga Pagpanilhig |RNA-Seq |ISO-seq |SLAF-seq
Ang Biomarker Technologies naghatag ug teknikal nga suporta sa Pacbio sequencing, Hi-C sequencing ug data analysis niini nga pagtuon.
Mga highlight
1. Ang unang chromosomal-level taas nga kalidad Rye genome nakuha, nga adunay usa ka chromosome gidak-on nga mas dako pa kay sa 1 Gb.
2.Kon itandi sa Tu, Aet ug Hv genome, usa ka talagsaon nga bag-o nga LTR-RT nga mga panghitabo ang naobserbahan sa Rye genome, nga maoy responsable sa pagpalapad sa rye genome nga gidak-on.
3. Ang divergence tali sa rye ug diploid trigo nahitabo human sa pagbulag sa sebada gikan sa trigo, uban sa divergence nga mga panahon alang sa duha ka mga panghitabo mao ang gibana-bana nga 9.6 ug 15 MYA.
Ang FT genes phosphorylation mahimong makontrol ang sayo nga kinaiya sa rye.
4.Selective sweep analysis nagpakita sa posibleng pag-apil sa ScID1 sa regulasyon sa petsa sa ulohan ug sa posibleng pagpili niini pinaagi sa domestication sa rye
Background
Background
Ang Rye kay usa ka bililhon nga pagkaon ug forage crop, usa ka importante nga genetic resource alang sa trigo ug triticale improvement, ug usa ka kinahanglanon nga materyal alang sa episyente nga comparative genomics nga mga pagtuon sa mga sagbot.Ang Weining rye, usa ka sayong namulak nga barayti nga gitanom sa China, talagsaon tungod sa lapad nga spectrum nga pagsukol niini sa powdery mildew ug stripe rust.Aron masabtan ang genetic ug molekular nga basehan sa mga elite nga kinaiya sa rye ug aron mapalambo ang genomic ug breeding nga mga pagtuon sa rye ug mga may kalabutan nga mga tanum, among gisunod-sunod ug gisusi ang genome sa Weining rye.
Mga nahimo
Rye Genome
Ang Rye genome natukod pinaagi sa pagsuklay sa mga basahon sa PacBio SMRT, mubo nga pagbasa sa Illumina sequencing, ingon man sa mga gikan sa chromatin conformation capture (Hi-C), genetic mapping, ug BioNano analysis.Ang gitigum nga mga contigs (7.74 Gb) nagkantidad sa 98.47% sa gibanabana nga gidak-on sa genome (7.86 Gb), nga adunay 93.67% sa mga contigs (7.25 Gb) nga gi-assign sa pito ka chromosome.Ang nagbalikbalik nga mga elemento naglangkob sa 90.31% sa gitigum nga genome.
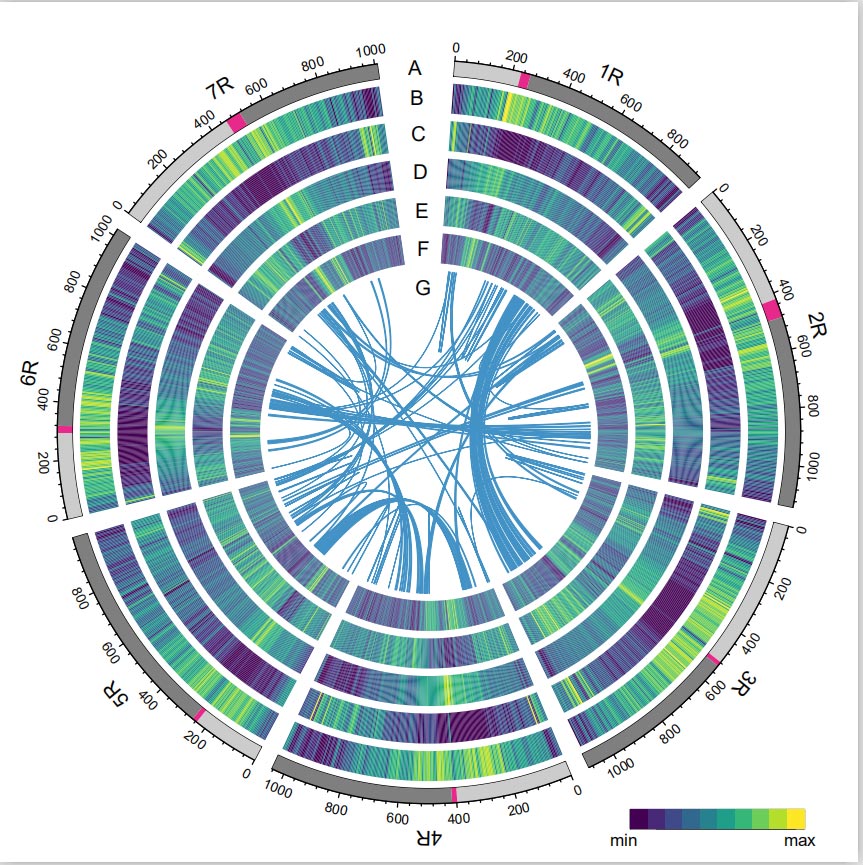
Rye Genome
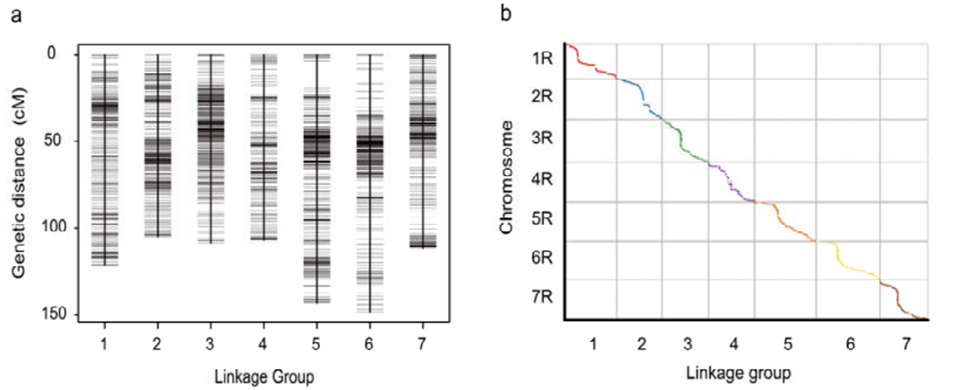
Ang genetic linkage map (WJ) naugmad gamit ang 295 F2 nga mga tanum nga nakuha gikan sa pagtabok sa duha ka rye landraces (Weining × Jingzhou)
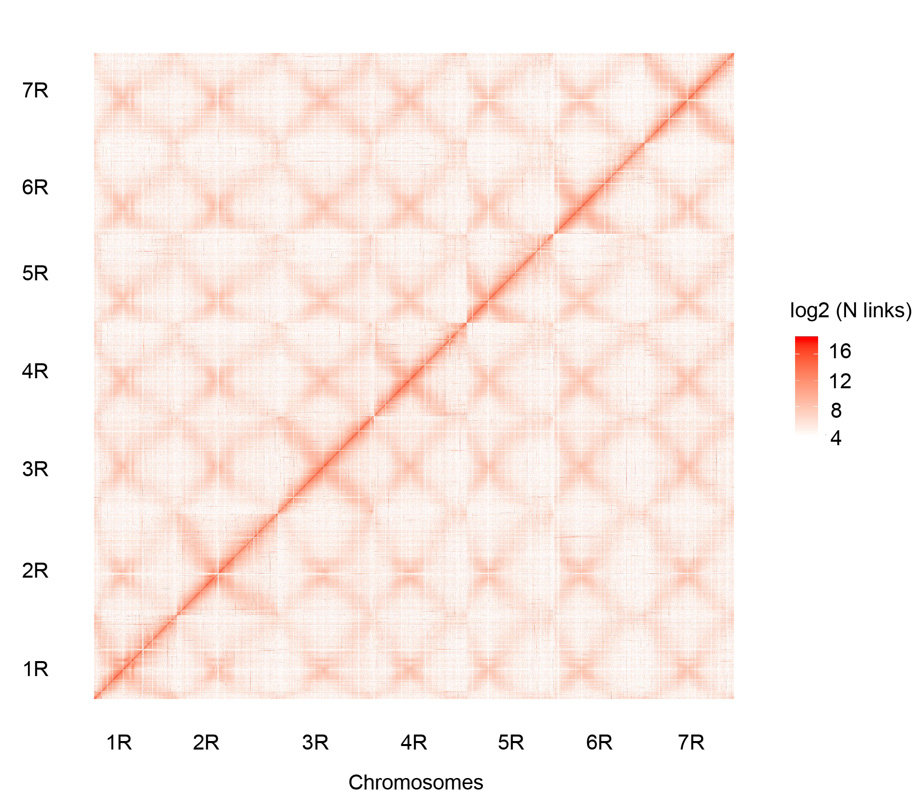
Mapa sa kontak sa Hi-C sa pito nga nagtipon nga Weining rye chromosome (1R - 7R)
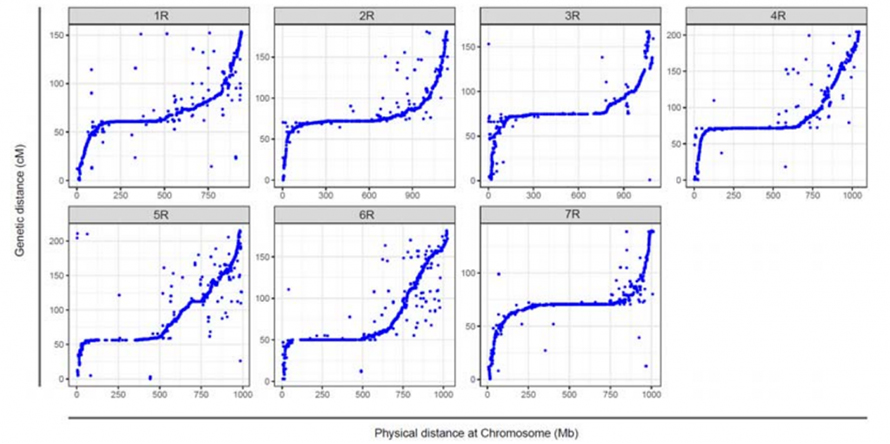
Ang pag-align tali sa pito ka mga assembled chromosome sa Weining rye ug ang pito ka rye linkage nga grupo naugmad gamit ang Lo7 x Lo255 RIL nga populasyon
Ang LTR Assembly Index (LAI) nga kantidad sa Rye genome nakit-an nga 18.42 ug 1,393 (96.74%) sa 1,440 nga gikonserbar kaayo nga BUSCO nga mga gene nahibal-an. Kini nga mga resulta nagsugyot nga ang Weining rye genome sequence adunay taas nga kalidad sa parehong intergenic. ug genic nga mga rehiyon.Usa ka kinatibuk-an nga 86,991 ka protina-coding genes, lakip ang 45,596 high-confidence(HC) genes ug 41,395 low-confidence genes (LC) genes ang gitagna.
2. Pagtuki sa mga TE
Pagtuki sa mga TE.Usa ka kinatibuk-an nga 6.99 Gb, nga nagrepresentar sa 90.31% sa Weining nga asembliya, gi-annotate isip TEs, nga naglakip sa 2,671,941 nga mga elemento nga iya sa 537 ka pamilya.Kini nga TE nga sulod klaro nga mas taas kaysa kaniadto nga gitaho alang sa Ta (84.70%), Tu (81.42%), Aet (84.40%), WEW (82.20%), o Hv (80.80%).Ang taas nga terminal repeat retrotransposons (LTR-RTs), lakip ang Gypsy, Copia ug unclassified RT nga mga elemento, mao ang dominanteng TEs, ug 1ccupied 84.49% sa annotated TE content ug 76.29 % sa assembled Weining genome;Ang mga transposon sa CACTA DNA mao ang ikaduha nga labing daghan nga TE, nga naglangkob sa 11.68% sa annotated nga TE content ug 10.55% sa gitigum nga Weining genome.
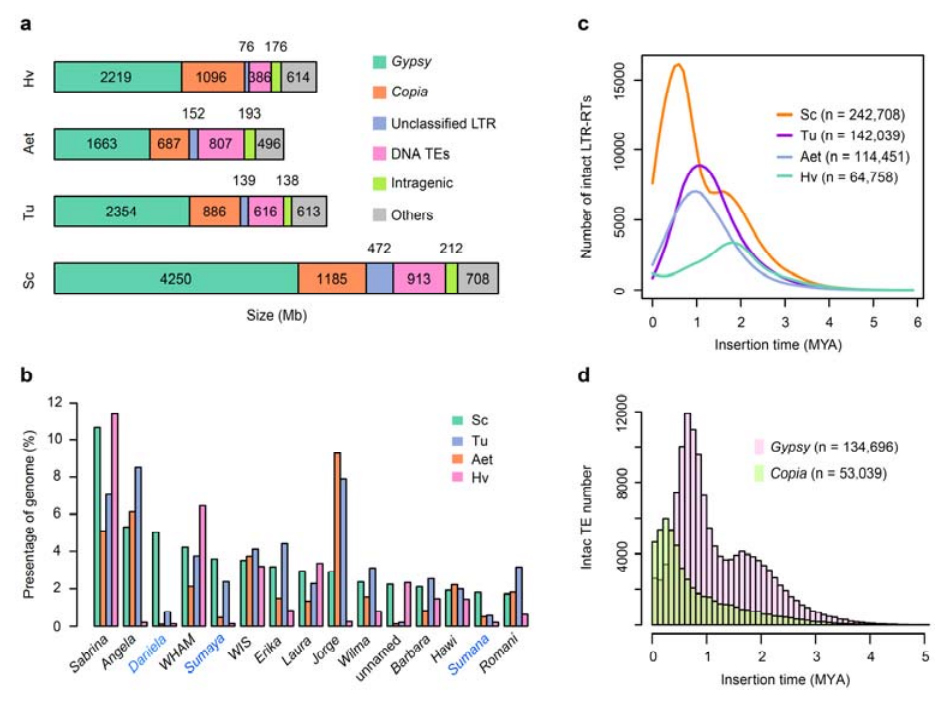
Pag-analisar sa mga elemento sa transposon sa rye
Ang Weining rye adunay medyo taas nga proporsiyon sa bag-ong mga pagsal-ot sa LTR-RTs nga ang kinapungkayan sa amplification nagpakita mga 0.5 milyon ka tuig na ang milabay (MYA), nga mao ang pinakabag-o sa upat ka espisye;ang laing peak, nahitabo gibana-bana nga 1.7 MYA, mas tigulang ug makita usab sa barley.Sa lebel sa superfamily, ang bag-o nga pagbuto sa mga elemento sa Copia sa Weining rye sa 0.3 MYA nakit-an, samtang ang mga amplification sa Gypsy RTs dominanteng naghulma sa bimodal distribution pattern sa LTR-RT burst dynamics.
3. Pagsusi sa rye genome evolution ug chromosome syntenies
Ang pagkalainlain tali sa rye ug diploid nga trigo nahitabo pagkahuman sa pagbulag sa sebada gikan sa trigo, nga ang mga oras sa pagkalainlain alang sa duha nga mga panghitabo nga gibana-bana nga 9.6 ug 15 MYA, matag usa.Ang 1R, 2R, 3R hingpit nga collinear sa mga grupo nga 1, 2 ug 3 nga mga chromosome sa trigo, matag usa.Ang 4R, 5R, 6R, 7R nakit-an nga adunay dagkong mga fusion ug mga bahin.
4. Pagtuki sa mga pagdoble sa gene ug ang epekto niini sa mga gene sa starch biosynthesis
Mamatikdan, ang gidaghanon sa mga tandemly duplicated genes (TDGs) ug proximally duplicated genes (PDGs) sa Weining rye parehas nga mas taas kaysa sa nakit-an para sa Tu, Aet, Hv, Bd ug Os.Ang mga transposed duplicated genes(TrDGs) mas daghan usab kay sa mga partikular nga nakit-an para sa Tu ug Aet.Ang pagpalapad sa genome sa Rye giubanan sa mas daghang gidaghanon sa mga pagdoble sa gene.Ang nadugangan nga pagbuto sa TE sa rye mahimong misangpot sa taas nga gidaghanon sa TrDGs.
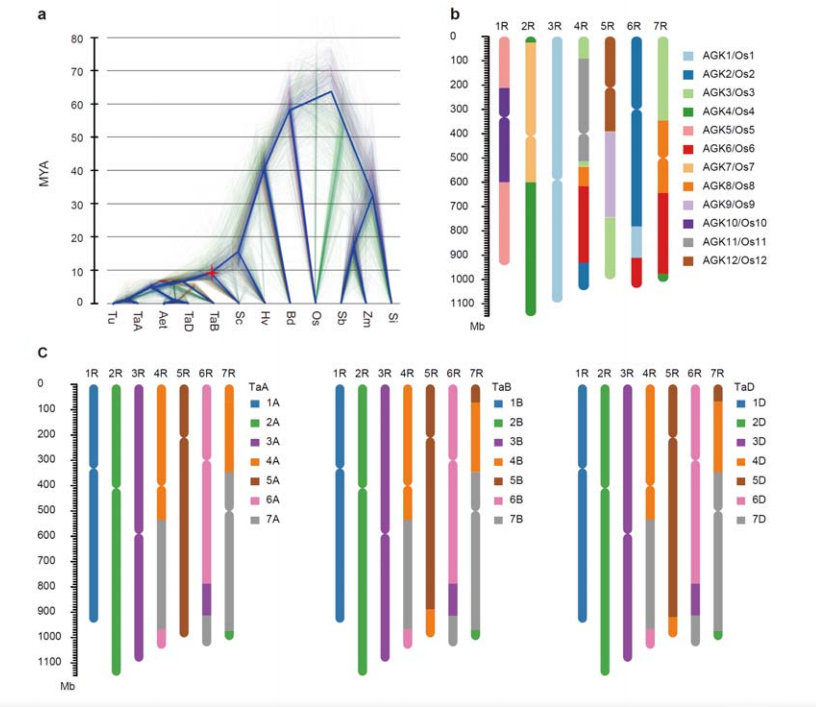
Pag-analisar sa ebolusyonaryo ug chromosome synteny sa rye genome
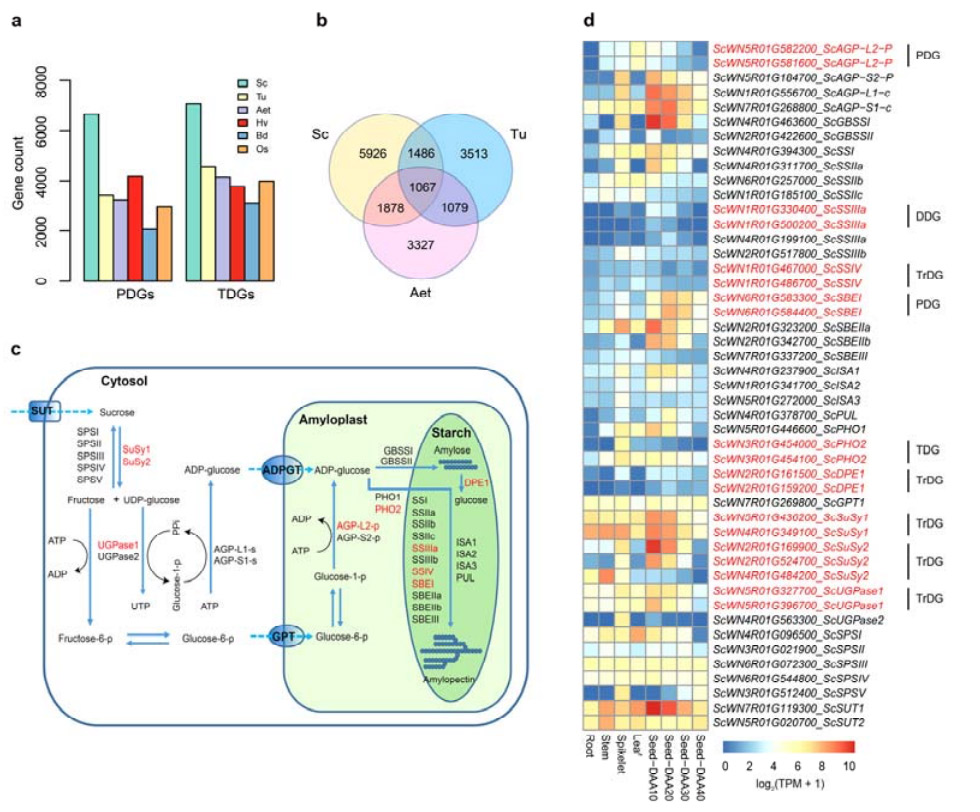
Pag-analisar sa mga duplikasyon sa rye gene ug ang epekto niini sa pagkalainlain sa mga gene nga may kalabutan sa biosynthesis sa starch (SBRGs)
5. Dissection sa rye seed storage protein (SSP) gene loci
Upat ka chromosomal loci (Sec-1 hangtod Sec-4) nga nagtino sa rye SSPs ang giila sa 1R o 2R.Ang mga gene nga α-gliadin bag-o lang nabag-o sa trigo ug sa mga espisye nga adunay suod nga relasyon pagkahuman sa pagkalainlain sa trigo gikan sa rye.
6. Pagsusi sa transcription factor (TF) ug mga gene nga resistensya sa sakit
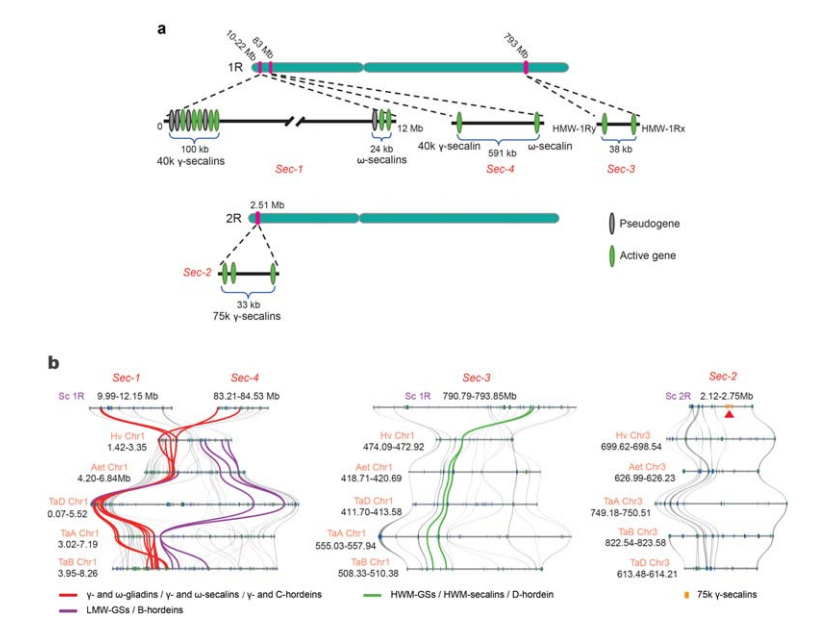
Pagtuki sa rye secalin loci
Ang Weining rye adunay mas daghang mga gene nga adunay kalabotan sa resistensya sa sakit (DRA) (1,989, Supplementary Data 3) kaysa Tu (1,621), Aet (1,758), Hv (1,508), Bd (1,178), Os (1,575), ug ang A (1,836). ), B (1,728) ug D (1,888) nga mga subgenome sa komon nga trigo.
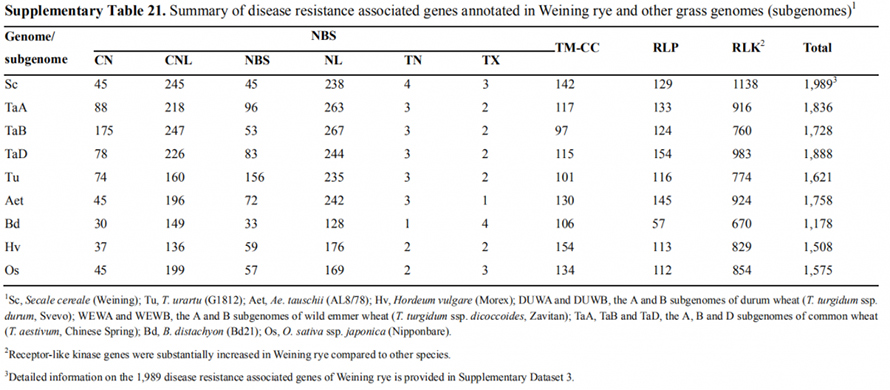
7. Pagsusi sa mga bahin sa ekspresyon sa gene nga nalangkit sa kinaiya sa sayo nga ulohan
Duha ka FT nga mga gene nga adunay medyo taas nga ekspresyon ubos sa taas nga mga kondisyon sa adlaw, ScFT1 ug ScFT2, gi-annotate sa Weining genome assembly.Duha ka amino acid residues sa ScFT2 (S76 ug T132) phosphorylation ang nakit-an nga relasyon sa pagpaubos sa kontrol sa oras
Mga bahin sa pag-uswag ug ekspresyon sa gene nga nalangkit sa sayo nga kinaiya sa Weining rye
8. Pagmina sa chromosomal regions ug loci nga posibleng nalangkit sa rye domestication
Kinatibuk-ang 123,647 ka SNP ang gigamit sa pagpahigayon ug selevtive sweep analysis tali sa kultibado nga rye ug S. vavilovii.11 ka pinili nga sweep signal nga giila sa reduction index (DRI), fixation index (FST) ug XP-CLR nga pamaagi.Ang ScID1 nakit-an nga posible nga pag-apil sa regulasyon sa petsa sa ulohan.
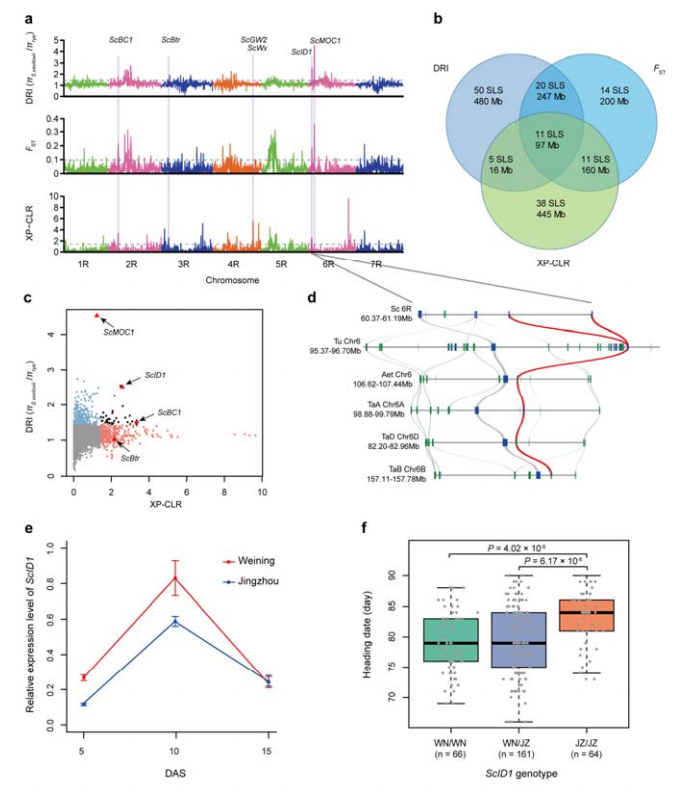
Pag-ila ug pagtuki sa mga rehiyon sa chromosomal ug loci nga posibleng may kalabutan sa pagpamuhi sa rye
Reperensya
Li GW ug uban pa.Ang usa ka taas nga kalidad nga genome assembly nagpasiugda sa rye genomic nga mga kinaiya ug agronomically importante nga mga gene.Nature Genetics (2021)
Mga Balita ug Highlight nagtumong sa pagpaambit sa pinakabag-o nga malampuson nga mga kaso sa Biomarker Technologies, pagkuha sa nobela nga mga kalampusan sa siyensya ingon man sa mga prominenteng teknik nga gigamit sa panahon sa pagtuon.
Oras sa pag-post: Ene-05-2022

