
Seqüenciació del transcriptoma complet - Illumina
Avantatges del servei
● Estimació de tot tipus d'ARN en termes de recomptes, expressió i expressió relativa basada en cromosomes
● Identificació d'ARN expressats diferencialment i expressió corresponent
● Anàlisi de coexpressió gènica
● Anàlisi de la xarxa ceRNA
● Anàlisi de vies implicades en gens clau
● Lliurament de resultats basat en BMKCloud: extracció de dades personalitzada disponible a la plataforma
● Serveis postvenda vàlids durant 3 mesos un cop finalitzat el projecte
Requisits de mostra i lliurament
| Biblioteca | Plataforma | Dades recomanades | QC de dades |
| ARN total | Illumina PE150 i SE50 | Circ/lnc/ARNm: 16G;miRNA: 10 milions de lectures | Q30≥85% |
Requisits de mostra:
Nucleòtids:
| Puresa | Integritat | Contaminació | Import |
| OD260/280≥1,7-2,5; OD260/230≥0,5-2,5; | Per a plantes: RIN≥6,5;Per a animals: RIN≥7;28S/18S≥1,0;elevació de referència limitada o nul·la | Es mostra al gel una contaminació limitada o nul·la de proteïnes o ADN. | Conc.≥100 ng/μl;Volum ≥ 10 μl;Total ≥ 2 μg |
Teixit: Pes (sec): ≥1 g
* Per a teixits de menys de 5 mg, recomanem enviar una mostra de teixit congelada (en nitrogen líquid).
Suspensió cel·lular: recompte de cèl·lules = 3×107
* Recomanem enviar lisat cel·lular congelat.En cas que el recompte de cèl·lules sigui inferior a 5 × 105, es recomana congelar ràpidament en nitrogen líquid.
Mostres de sang:
PA×genBloodRNATube;
6 ml de TRIzol i 2 ml de sang (TRIzol: sang = 3: 1)
Lliurament de mostres recomanat
Contenidor:
Tub de centrífuga de 2 ml (no es recomana paper d'alumini)
Etiquetatge de la mostra: grup+rèplica, p. ex. A1, A2, A3;B1, B2, B3....
Enviament:
1.Gel sec: les mostres s'han d'envasar en bosses i enterrar-les en gel sec.
2. Tubs RNAstable: les mostres d'ARN es poden assecar en un tub d'estabilització d'ARN (per exemple, RNAstable®) i enviar-se a temperatura ambient.
Flux de treball del servei

Disseny d'experiments

Lliurament de la mostra

Extracció d'ARN

Construcció de la biblioteca

Seqüenciació

Anàlisi de dades

Serveis postvenda
Bioinformàtica
1. Estimació de tot tipus d'ARN basada en l'expressió relativa
Circos sobre la importància de les diferències en l'expressió d'ARN
2. Xarxa de vies KEGG integrada
Xarxa integrada de vies KEGG
3.Anàlisi de la xarxa ceRNA
ceRNA Interaccions DE-circRNA-miRNA-mRNA basades en xarxa
Cas BMK
Patró d'expressió de CircRNA i xarxes ceRNA i miRNA-mRNA implicades en el desenvolupament d'anteres a la línia CMS de Brassica campestris
Publicat:Revista Internacional de Ciències Moleculars,2019
Les cèl·lules KP (shRNA-2) i el control negatiu (sh-Scr) es van obtenir el dia 6 d'una infecció viral específica.
Resultats clau
Aquest estudi va establir la línia d'esterilitat masculina (CMS) del citoplasma de Polima "Bcpol97-05A" i la línia fèrtil, "Bcajh97-01B", a Brassica campestris L. ssp.chinensis Makino, syn.B. rapa ssp.chinensis i va realitzar comparacions de perfils d'expressió d'ARN entre els brots florals de la línia estèril i la línia fèrtil mitjançant la seqüenciació del transcriptoma sencer.
1. Es van identificar un total de 31 circRNAs expressats de manera diferencial (DE), 47 DE miRNAs i 4779 DE mRNAs.
2. L'anàlisi d'ontologia gènica (GO) va demostrar que la majoria dels gens DE estaven implicats en el desenvolupament de la paret del pol·len
3.L'anàlisi de l'enriquiment de la via KEGG dels ARNm DE (inclosos els ARNm regulats a l'alça i a la baixa a la línia estèril en comparació amb la línia fèrtil) va demostrar que el "metabolisme del midó i la sacarosemia", la "biosíntesi de fenilpropanoides" i les "interconversions de pentosa i glucuronat". ” eren les vies metabòliques més enriquides.
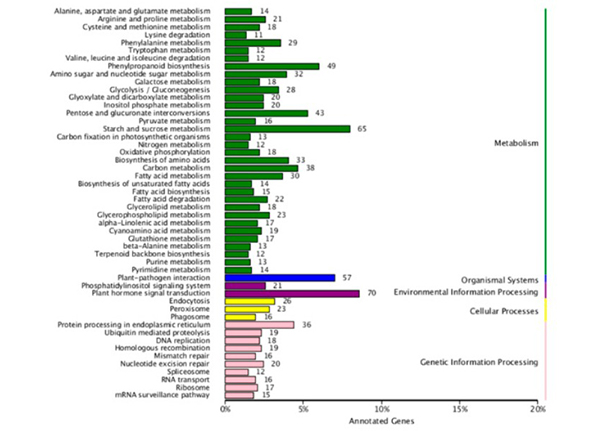 Anàlisi d'enriquiment de la via KEGG dels ARNm expressats de manera diferencial | 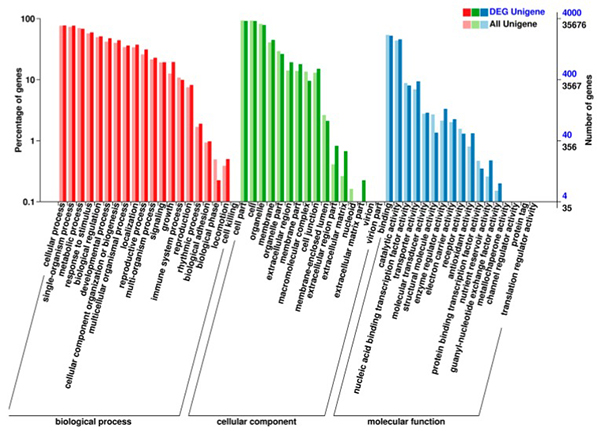 Classificació d'ontologia gènica (GO) dels ARNm expressats de manera diferencial | 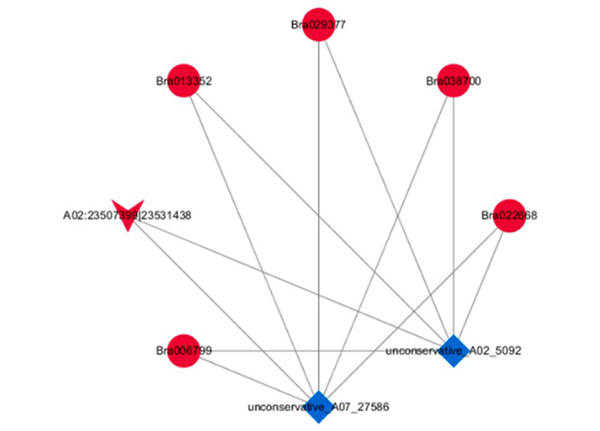 Vista de la xarxa triple DEcircRNA–DEmiRNA–DEmRNA |
Referència
Liang Y , Zhang Y , Xu L , et al.Patró d'expressió de CircRNA i xarxes ceRNA i miRNA-mRNA implicades en el desenvolupament d'anteres a la línia CMS de Brassica campestris [J].Revista Internacional de Ciències Moleculars, 2019, 20(19):4808-.DOI: 10.3390/ijms20194808














