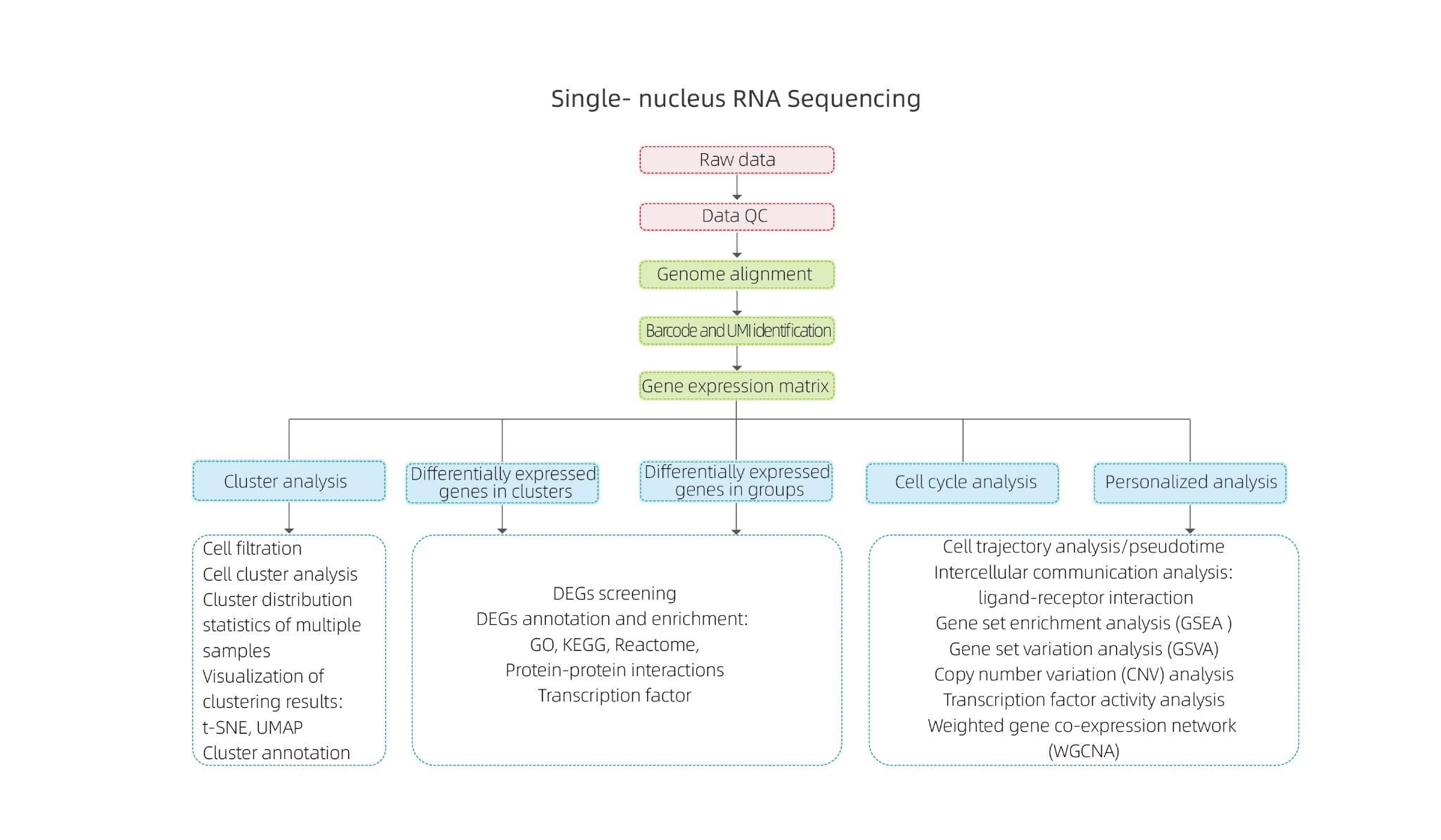
Seqüenciació d'ARN d'un sol nucli
Principi tècnic
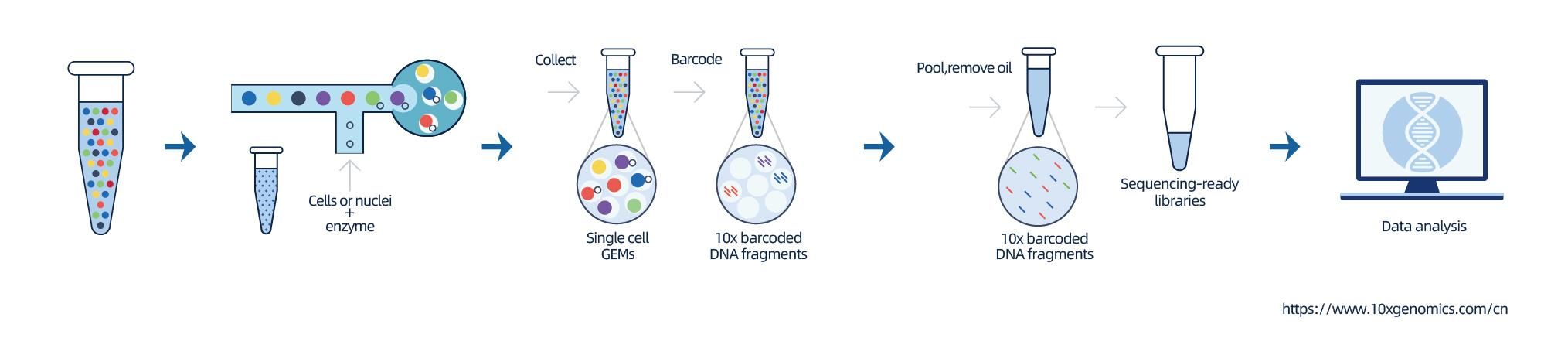
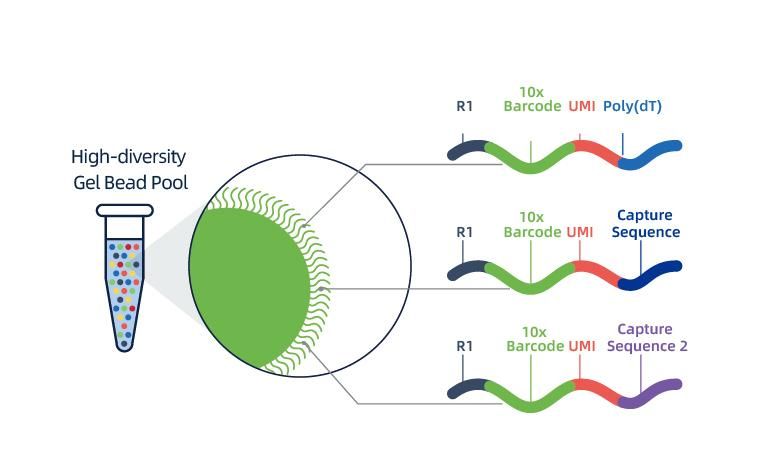
L'aïllament dels nuclis s'aconsegueix mitjançant 10× Genomics ChromiumTM, que consta d'un sistema de microfluídica de vuit canals amb dobles encreuaments.En aquest sistema, s'encapsulen perles de gel amb codis de barres i imprimació, enzims i un únic nucli en una gota d'oli de la mida d'un nanolitre, generant perles de gel en emulsió (GEM).Un cop formats els GEM, es realitza la lisi cel·lular i l'alliberament de codis de barres a cada GEM.L'ARNm es transcriu inversament en molècules d'ADNc amb codis de barres 10 × i UMI, que estan subjectes a més a la construcció estàndard de la biblioteca de seqüenciació.
El teixit no és adequat per a la preparació de suspensió cel·lular
| Cèl·lula / Teixit | Raó |
| Teixit congelat no fresc | No es poden obtenir organitzacions noves o desades des de fa temps |
| Cèl·lula muscular, megacariocit, greix... | El diàmetre de la cel·la és massa gran per entrar a l'instrument |
| fetge… | Massa fràgil per trencar-se, incapaç de distingir cèl·lules individuals |
| Cèl·lula neuronal, cervell... | Més sensible, fàcil d'estressar, canviarà els resultats de la seqüenciació |
| Pàncrees, tiroides... | Ric en enzims endògens, que afecten la producció de suspensió unicel·lular |
Nucli únic vs unicel·lular
| Nucli únic | Unicel·lular |
| Diàmetre cel·lular il·limitat | Diàmetre cel·lular: 10-40 μm |
| El material pot ser teixit congelat | El material ha de ser teixit fresc |
| Baix estrès de les cèl·lules congelades | El tractament enzimàtic pot provocar una reacció d'estrès cel·lular |
| No cal eliminar glòbuls vermells | Cal eliminar els glòbuls vermells |
| Nuclear expressa bioinformació | Tota la cèl·lula expressa bioinformació |
Especificacions del servei
| Biblioteca | Estratègia de seqüenciació | Volum de dades | Exemples de requisits | Teixit |
| 10× Biblioteca de nuclis únics de genòmica | 10x Genomics -Illumina PE150 | 100.000 lectures/cel·la aprox.100-200 Gb | Número de cel·la: > 2 × 105 Cel·lular conc.a 700-1.200 cèl·lules/μL | ≥ 200 mg |
Per obtenir més detalls sobre la guia de preparació de mostres i el flux de treball del servei, no dubteu a parlar amb aExpert en BMKGENE
Flux de treball del servei

Disseny d'experiments

Lliurament de la mostra
Aïllament de nuclis

Construcció de la biblioteca

Seqüenciació

Anàlisi de dades

Serveis postvenda
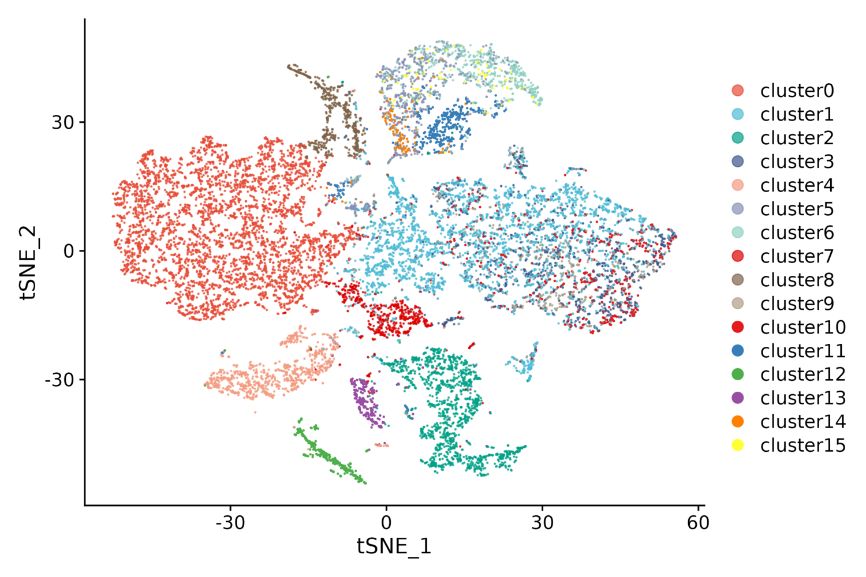
1.Agrupació puntual
2.Mapa de calor de agrupació d'abundància d'expressió del marcador
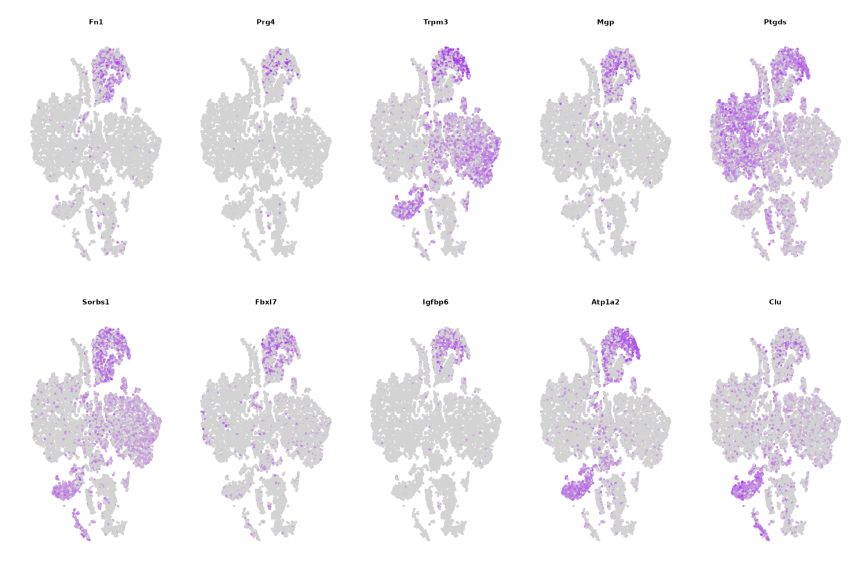
 3.Distribució del gen Maker en diferents clústers
3.Distribució del gen Maker en diferents clústers
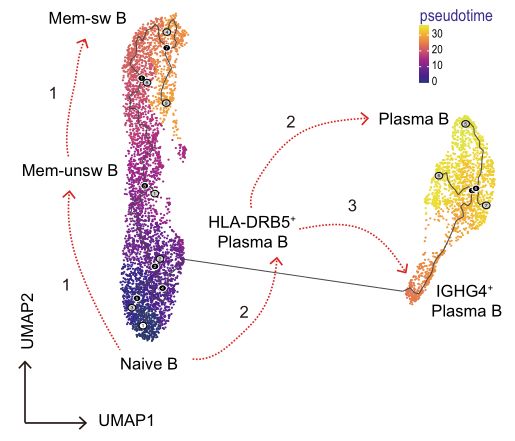
4.Anàlisi de la trajectòria cel·lular/pseudotemps