GENÒMICA HUMANA
genètica de la natura
La seqüenciació de lectura llarga identifica les expansions repetides de GGC a NOTCH2NLC associades a la malaltia d'inclusió intranuclear neuronal
ONT reseqüenciació |Il·lumina |Seqüenciació de l'exoma sencer |Seqüenciació dirigida a CRISPR-Cas9 ONT |ARN-seq |Crida de metilació ONT 5mC
Destacats
1. Mitjançant l'anàlisi d'enllaços en una gran família de NIID, es van identificar dues regions enllaçades.
La seqüenciació de lectura llarga basada en ONT i la seqüenciació ONT d'enriquiment mediat per Cas-9 van descobrir una possible causa genètica de les expansions de repetició de NIID, GGC en 5′ UTR de NOTCH2NLC.Aquest estudi va informar per primera vegada d'expansions repetides en gens específics per a humans que van evolucionar mitjançant duplicacions segmentàries.
3.La seqüenciació de l'ARN va revelar transcripcions antisentit anormals al principi o dins de les regions d'expansió de repetició de GGC a NOTCH2NLC.
Fons
NLa malaltia d'inclusió intranuclear euronal (NIID) és una malaltia neurodegenerativa progressiva i mortal, que es caracteritza per la presència d'inclusions intranuclears hialines eosinofíliques al sistema nerviós central i perifèric.Les seves manifestacions clíniques molt variables plantegen grans dificultats en el diagnòstic fins a la introducció de la biòpsia cutània.Tanmateix, els mètodes basats en histopatologia encara pateixen un diagnòstic errònia, que demana una comprensió genètica de la NIID.
Assoliments
Anàlisi d'enllaços
SLa seqüenciació hort-read basada en la seqüenciació del genoma sencer (WGS) i la seqüenciació completa de l'exoma (WES) es va realitzar en una gran família de NIID (13 membres afectats i 7 no afectats).L'anàlisi d'enllaços dels SNP extrets d'aquestes dades només va revelar dues regions enllaçades: una regió de 3,5 Mb a 1p36.31-p36.22 (LOD màxim = 2,32) i una regió de 58,1 Mb a 1p22.1-q21.3 (LOD màxim: 4,21). ).Tanmateix, no es van identificar SNP o CNV patògens en aquestes regions enllaçades.
GGC repeteix les expansions a NOTCH2NLC
NLa seqüenciació basada en anopores es va processar en 13 membres afectats i 4 no afectats de 8 famílies (un altre membre afectat va ser seqüenciat per la plataforma de seqüenciació de lectura llarga Pacbio).Les dades de lectura llarga van revelar expansions repetides de GGC associades a malalties en el mapeig del gen 5′ UTR de NOTCH2NLC a la regió enllaçada de 58, 1 Mb (figura 1).Aquestes expansions repetides també es van identificar en els 40 casos esporàdics de NIID provats per RP-PCR.
CLa seqüenciació d'objectius mediada per as-9 a la plataforma de nanopores es va utilitzar per aconseguir una major cobertura de lectura a la repetició NOTCH2NLC (100 X-1.795 X).Aquestes seqüències de consens van coincidir bé amb les troballes anteriors sobre les expansions repetides de GGC.A més, es van identificar les repeticions {(GGA)n (GGC)n}n com a marcador genètic potencial per al fenotip dominant de debilitat (figura 2).
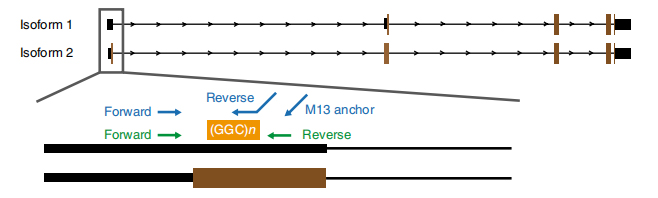
Figura 1. Expansió repetida associada a la malaltia identificada a l'exó 1 de les isoformes NOTCH2NLC.
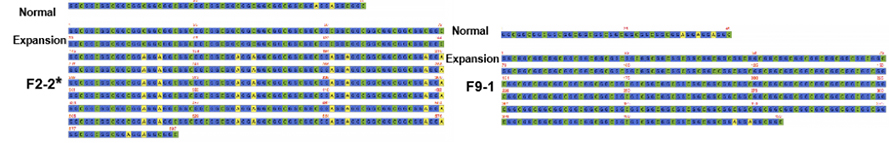
Figura 2. Seqüències consensuades de repetició de NPTCH2NLC en pacients amb NIID amb (*) o sense fenotip dominant de debilitat
NEls gens OTCH2NL són gens específics per a humans, que es creu que tenen un paper vital en l'evolució del cervell humà i les malalties neurològiques.Tanmateix, tres gens relacionats amb NOTCH2 (NOTCH2NLA, NOTCH2NLB i NOTCH2NLC) amb una identitat de seqüència > 99, 1% no es van resoldre fins a l'últim muntatge del genoma humà.La seqüenciació sense síntesi i de lectura llarga a la plataforma de nanopors ha mostrat avantatges notables en la resolució de regions d'alta similitud i repeticions (GGC)n amb 100% rics en GC.
GGC repeteix les expansions a NOTCH2NLC
TLa seqüenciació del ranscriptoma es va processar en 2 membres afectats i 2 no afectats.Es va calcular la profunditat de lectura normalitzada a les cadenes de sentit i antisentit a aigües amunt dels primers exons dels paràlegs NOTCH2NL.Només es van trobar transcripcions antisens anormals en casos afectats, que es troben al principi o dins de la regió d'expansió repetida (pics morats a F1-14 i F1-16 a la figura 3).A més, es van identificar 54 DEG i tots es van enriquir en termes GO i MPO relacionats amb les funcions neuronals.
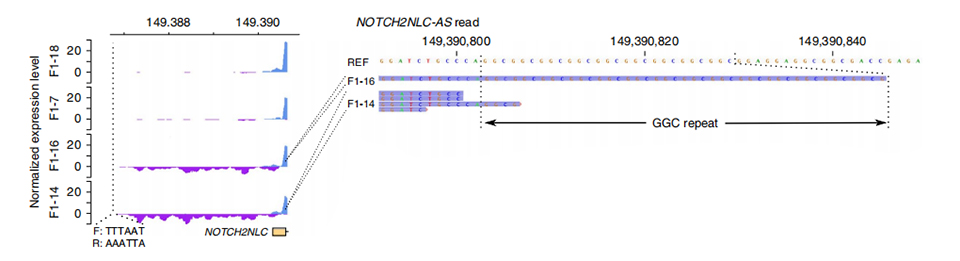
Figura 3. Profunditat de lectura normalitzada aigües amunt del primer exó de NOTCH2NLC en casos no afectats (a dalt) i afectats (a sota).
Tecnologia
Oxford Nanopore Teghnologies (ONT)
NLa seqüenciació d'anopores es distingeix d'altres plataformes de seqüenciació, ja que els nucleòtids es llegeixen directament sense procés de síntesi d'ADN.Quan l'ADN d'una sola cadena passa a través d'un porus proteic de mida nanomètrica (nanopor), diferents nucleòtids generen diferents corrents iònics, que es poden capturar i transferir a una seqüència de bases.La pròpia plataforma de seqüenciació ONT no mostra un límit tècnic aparent sobre la durada de la lectura de l'ADN.Per tant, les lectures ultra llargues (ULR) estan disponibles per al muntatge del genoma d'alta qualitat.A més, aquestes lectures extremadament llargues, prou llargues per creuar característiques de seqüències complexes o variacions estructurals, ajuden a superar les limitacions de la seqüenciació de lectura curta aquí.

Seqüenciació de nanopors
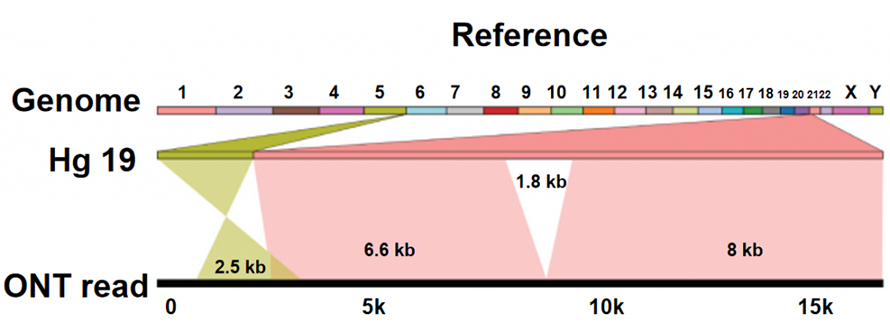
Identificació de la variació d'estructura (SV).
SLa seqüenciació sense síntesi va preservar en gran mesura la informació de metilació de l'ADN a la plantilla.A, T, C i G metilats generen corrents iònics diferents dels no metilats, que es poden llegir directament per la plataforma.La seqüenciació de nanoporos permet el perfil del genoma sencer de 5mC i 6mA amb resolució d'un sol nucleòtid.
Referència
Jun Sone, et.al.La seqüenciació de lectura llarga identifica les expansions repetides de GGC a NOTCH2NLC associades a la malaltia d'inclusió intranuclear neuronal.Genètica de la natura (2019)
Tecnologia i aspectes destacats té com a objectiu compartir l'aplicació més recent amb èxit de diferents tecnologies de seqüenciació d'alt rendiment en diversos àmbits de recerca, així com idees brillants en disseny experimental i mineria de dades.
Hora de publicació: 06-gen-2022

