TRANSCRIPTOMICA
naturalesa
COMUNICACIONS
La caracterització de la transcripció completa de la mutació SF3B1 en la leucèmia limfocítica crònica revela la regulació a la baixa dels introns retinguts
Transcripcions integrals|Seqüenciació de nanopors|Anàlisi d'isoformes alternatives
Fons
SS'ha informat àmpliament que les mutacions omàtiques en el factor d'splicing SF3B1 s'associen amb diversos càncers, com ara la leucèmia limfocítica crònica (LLC), el melanoma uveal, el càncer de mama, etc.Tanmateix, els estudis sobre aquests patrons d'empalmament alternatius s'han limitat durant molt de temps al nivell d'esdeveniment i a la manca de coneixement a nivell d'isoforma a causa de la limitació de les transcripcions reunides de lectura curta.Aquí, es va introduir la plataforma de seqüenciació de nanopors per generar transcripcions de longitud completa, que van potenciar la inverstigació a les isoformes AS.
Disseny experimental
Experiments
Agrupació:1. CLL-SF3B1(WT) 2. CLL-SF3B1 (mutació K700E);3. Cèl·lules B normals
Estratègia de seqüenciació:Seqüenciació de la biblioteca MinION 2D, seqüenciació de la biblioteca PromethION 1D;dades de lectura curta de les mateixes mostres
Plataforma de seqüenciació:ONT Minion;ONT PromethION;
Anàlisi Bioinformàtica
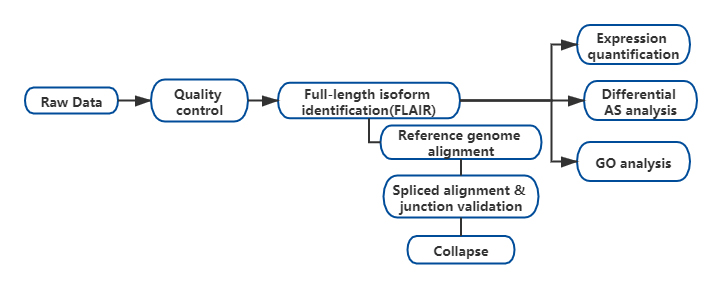
Resultats
AEs van generar un total de 257 milions de lectures a partir de 6 mostres de CLL i 3 cèl·lules B.De mitjana, el 30,5% d'aquestes lectures es van identificar com a transcripcions completes.
FEs va desenvolupar una anàlisi d'isoformes alternativa de llargària completa de l'ARN (FLAIR) per generar un conjunt d'isoformes d'alta confiança.FLAIR es pot resumir com:
Nanopore llegeix l'alineació: identifica l'estructura general de la transcripció basada en el genoma de referència;
SCorrecció d'unió de plices: corregeix errors de seqüència (vermell) amb el lloc d'empalmament d'introns anotats, introns de dades de lectura curta o ambdós;
Collapse: resumeix isoformes representatives basades en cadenes d'unió d'empalmament (conjunt de primer pas).Seleccioneu una iso de confiança alta en funció del nombre de lectures compatibles (Llindar: 3).
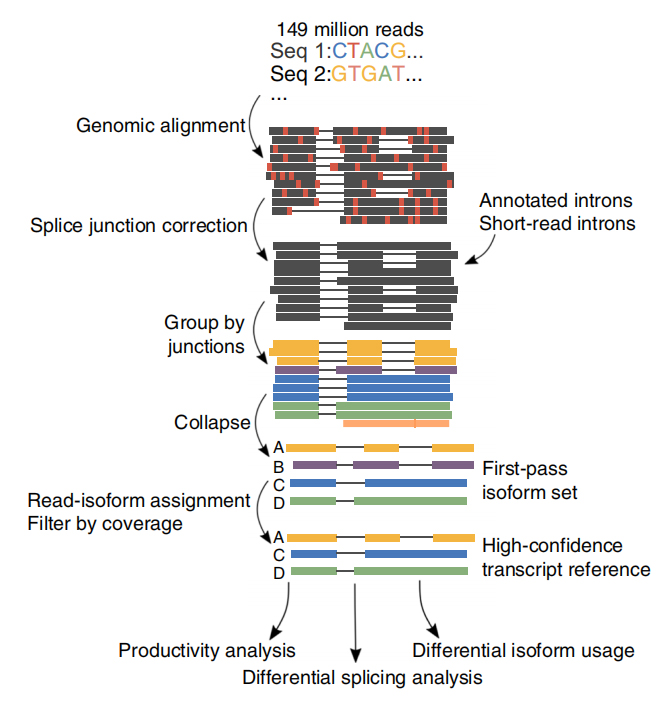
Figura 1. Anàlisi FLAIR per identificar isoformes de longitud completa associades a la mutació de SF3B1 en CLL
FLAIR va identificar 326.699 isoformes empalmades d'alta confiança, el 90% de les quals són isoformes noves.Es va trobar que la majoria d'aquestes isoformes no anotades eren noves combinacions d'unions d'empalmament conegudes (142.971), mentre que la resta d'isoformes noves contenien intró retingut (21.700) o nou exó (3594).
Lles seqüències de lectura prolongada permeten la identificació de llocs d'empalmament alterats per SF3B1-K700E mutants a nivell d'isoforma.Es va trobar que 35 alternatives 3'SS i 10 alternatives 5'SS es van empalmar de manera significativament diferencial entre SF3B1-K700E i SF3B1-WT.33 de les 35 alteracions van ser descobertes recentment per seqüències de lectura llarga.A les dades de Nanopore, la distribució de la distància entre els 3'SS alterats per SF3B1-K700E als pics dels llocs canònics és d'uns -20 pb, que difereix significativament d'una distribució de control, similar al que s'ha informat a les seqüències de lectura curta de CLL.Es van analitzar les isoformes del gen ERGIC3, on es va trobar una nova isoforma que contenia el lloc d'empalmament proximal més abundant a SF3B1-K700E.Tant el 3'SS proximal com el distal es van associar amb patrons d'AS diferents que generaven múltiples isoformes.
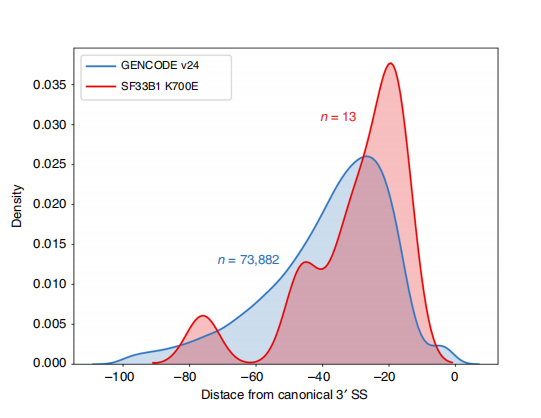
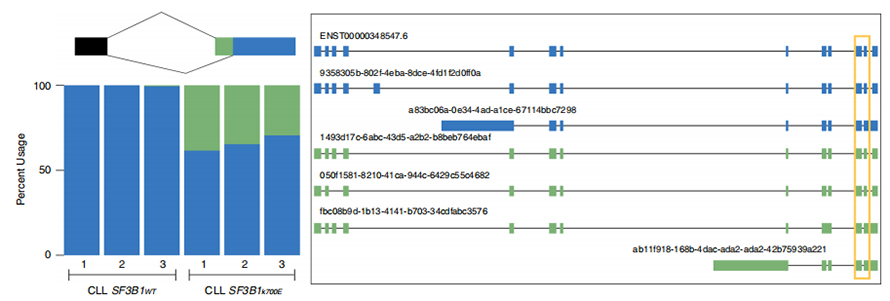
Figura 2. Patrons alternatius d'empalmament de 3′ identificats amb dades de seqüenciació de nanoporos
L'anàlisi de l'ús d'esdeveniments d'IR ha estat limitada durant molt de temps en l'anàlisi basada en lectura curta a causa de la confiança en la identificació i quantificació d'IR.L'expressió de les isoformes IR a SF3B1-K700E i SF3B1-WT es va quantificar a partir de seqüències de nanopors, revelant una regulació global a la baixa de les isoformes IR a SF3B1-K700E.
Figura 4. Intensitat agrícola i connectivitat de xarxa a través de tres sistemes agrícoles (A i B);Anàlisi forestal aleatòria (C) i relació entre la intensitat agrícola i la colonització d'AMF (D)
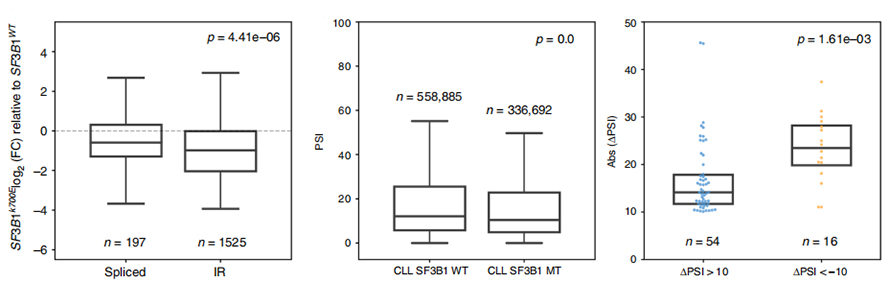
Figura 3. Els esdeveniments de retenció d'introns estan més fortament regulats a la baixa a CLL SF3B1-K700E
Tecnologia
Seqüenciació de lectura llarga Nanopore
NLa seqüenciació d'anopores és una tecnologia de seqüenciació de senyal elèctric en temps real d'una sola molècula.
DL'ADN o ARN de doble cadena s'uneix a la proteïna nanoporosa incrustada al biofilm i es desenrotlla sota el lideratge de la proteïna motora.
DLes cadenes de NA/ARN passen a través de la proteïna del canal de nanoporos a una velocitat determinada sota l'acció de la diferència de voltatge.
Mles olècules generen diferents senyals elèctrics segons l'estructura química.
Rla detecció en temps real de seqüències s'aconsegueix mitjançant la trucada de base.
Rendiment de la seqüenciació de transcriptoma de llarga durada
√ Saturació de dades
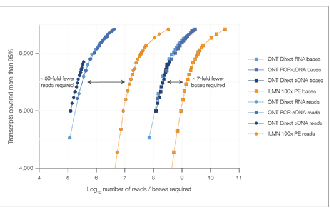
Es necessiten 7 vegades menys lectures per aconseguir una saturació de dades comparable.
√ Identificació de l'estructura de la transcripció
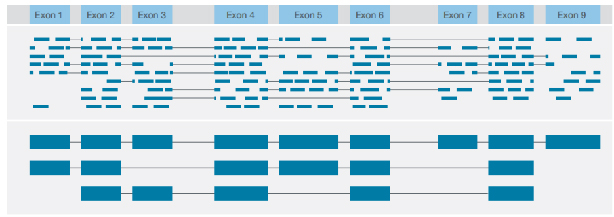
Identificació de diverses variants estructurals amb lectura completa consensuada de cada transcripció
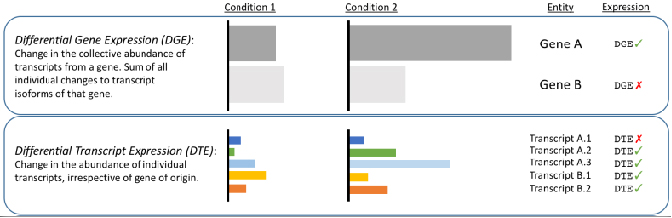
√ Anàlisi diferencial a nivell de transcripció -Revelar canvis ocults per lectures curtes
Referència
Tang AD , Soulette CM , Baren MJV , et al.La caracterització de la transcripció completa de la mutació SF3B1 en la leucèmia limfocítica crònica revela la baixada dels introns retinguts [J].Comunicacions de la natura.
Tecnologia i aspectes destacats té com a objectiu compartir l'aplicació més recent amb èxit de diferents tecnologies de seqüenciació d'alt rendiment en diversos àmbits de recerca, així com idees brillants en disseny experimental i mineria de dades.
Hora de publicació: 08-gen-2022

