
Секвениране на целия транскриптом – Illumina
Предимства на услугата
● Оценка на всички видове РНК по отношение на броя, експресията и относителната експресия на базата на хромозома
● Идентифициране на диференциално експресирани РНК и съответстваща експресия
● Анализ на генна ко-експресия
● ceRNA мрежов анализ
● Анализ на пътя на ключовите гени
● Предоставяне на резултати, базирано на BMKCloud: Персонализирано извличане на данни, достъпно на платформата
● Следпродажбено обслужване, валидно за 3 месеца след завършване на проекта
Примерни изисквания и доставка
| Библиотека | Платформа | Препоръчителни данни | Данни QC |
| Обща РНК | Illumina PE150&SE50 | Circ/lnc/mRNA: 16G;miRNA: 10 милиона четения | Q30≥85% |
Примерни изисквания:
Нуклеотиди:
| Чистота | Интегритет | Замърсяване | Количество |
| OD260/280≥1,7-2,5; OD260/230≥0,5-2,5; | За растения: RIN≥6.5;За животни: RIN≥7;28S/18S≥1.0;ограничено или никакво повишение на базовата линия | Ограничено или никакво протеиново или ДНК замърсяване, показано на гела. | Конц.≥100 ng/μl;Обем ≥ 10 μl;Общо ≥ 2 μg |
Кърпа: Тегло (суха): ≥1 g
*За тъкани, по-малки от 5 mg, препоръчваме да изпратите бързо замразена (в течен азот) тъканна проба.
Клетъчна суспензия: Брой клетки = 3×107
*Препоръчваме да изпратите замразен клетъчен лизат.В случай, че броят на клетките е по-малък от 5 × 105, се препоръчва бързо замразяване в течен азот.
Кръвни проби:
PA×geneBloodRNATube;
6mLTRIzol и 2mL кръв (TRIzol:Кръв=3:1)
Препоръчителна доставка на мостри
Контейнер:
2 ml центрофужна епруветка (не се препоръчва калаено фолио)
Примерно етикетиране: Групиране+репликат напр. A1, A2, A3;B1, B2, B3... ...
Пратка:
1. Сух лед: Пробите трябва да бъдат опаковани в торби и заровени в сух лед.
2. РНК стабилни епруветки: РНК пробите могат да бъдат изсушени в РНК стабилизираща епруветка (напр. RNAstable®) и изпратени при стайна температура.
Работен поток на услугата

Дизайн на експеримента

Доставка на мостри

Екстракция на РНК

Изграждане на библиотека

Секвениране

Анализ на данни

Следпродажбени услуги
Биоинформатика
1. Оценка на всички видове РНК базирани на относителна експресия
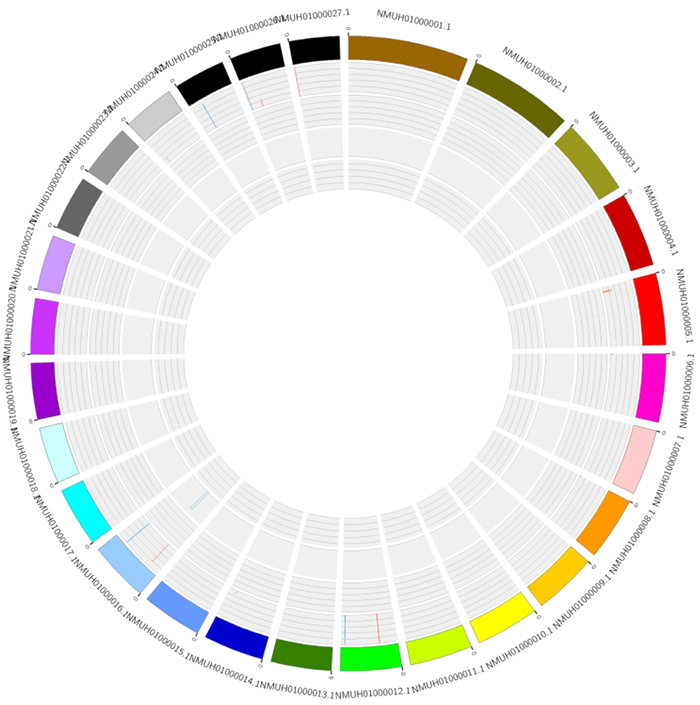
Circos за значимостта на разликите в експресията на РНК
2.Интегрирана мрежа от пътища на KEGG
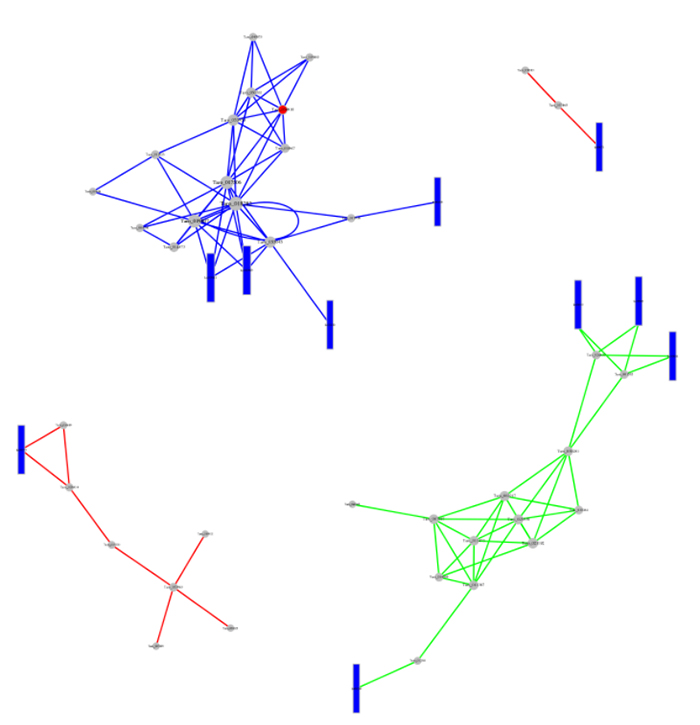
Интегрирана мрежа от пътища на KEGG
3. ceRNA мрежов анализ
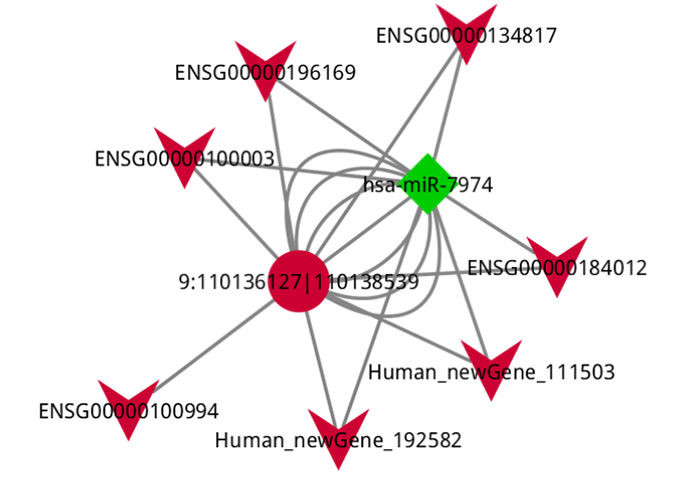
ceRNA Мрежово базирани DE-circRNA-miRNA-mRNA взаимодействия
Калъф BMK
Модел на експресия на CircRNA и ceRNA и miRNA-mRNA мрежи, участващи в развитието на прашника в CMS линията на Brassica campestris
Публикувано:Международен журнал за молекулярни науки,2019 г
NONMMUT015812-нокдаун KP (shRNA-2) клетки и отрицателни контролни (sh-Scr) клетки бяха получени на ден 6 от специфична вирусна инфекция.
Ключови резултати
Това проучване установява линията за мъжка стерилност (CMS) на Polima цитоплазма „Bcpol97-05A“ и фертилната линия „Bcajh97-01B“ в Brassica campestris L. ssp.chinensis Makino, син.B. rapa ssp.chinensis и извършиха сравнения за профилиране на РНК експресия между цветните пъпки на стерилната линия и фертилната линия чрез секвениране на целия транскриптом.
1. Идентифицирани са общо 31 диференциално експресирани (DE) circRNA, 47 DE miPHK и 4779 DE mRNA.
2. Анализът на генната онтология (GO) показа, че повечето от DE гените са участвали в развитието на поленовите стени
3. Анализът на обогатяване на пътя на KEGG на DE иРНК (включително регулираните нагоре и надолу иРНК в стерилната линия в сравнение с фертилната линия) показа, че „метаболизъм на нишесте и захароза“, „биосинтеза на фенилпропаноид“ и „взаимни преобразувания на пентоза и глюкуронат ” бяха най-обогатените метаболитни пътища.
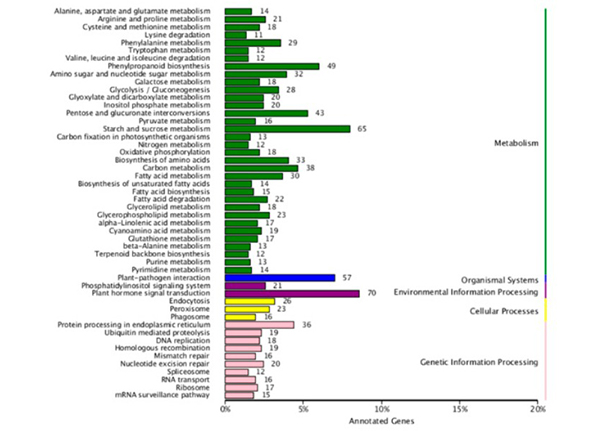 Анализ на обогатяване на пътя на KEGG на диференциално експресираните иРНК | 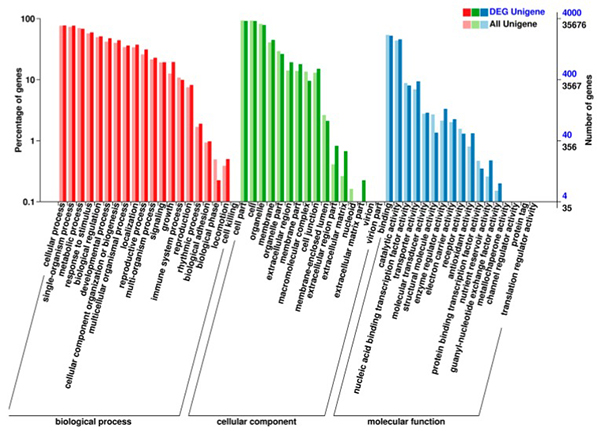 Класификация на генната онтология (GO) на диференциално експресираните иРНК | 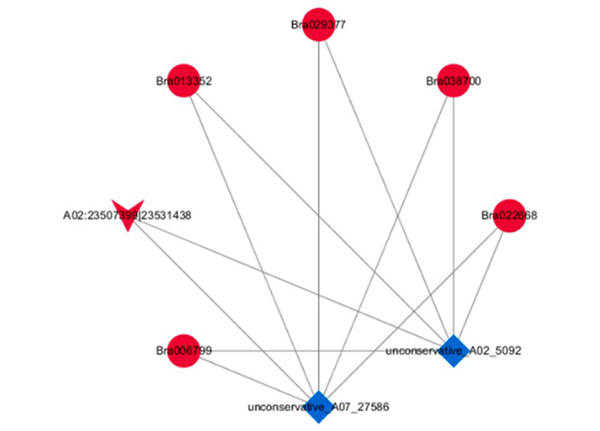 Изглед на тройната мрежа DEcircRNA–DEmiRNA–DEmRNA |
справка
Liang Y, Zhang Y, Xu L и др.Модел на експресия на CircRNA и ceRNA и miRNA-mRNA мрежи, участващи в развитието на прашника в CMS линията на Brassica campestris [J].Международен журнал за молекулярни науки, 2019, 20 (19): 4808-.DOI: 10.3390/ijms20194808














