ТРАНСКРИПТОМИКА
природа
КОМУНИКАЦИИ
Характеризиране на препис с пълна дължина на SF3B1 мутация при хронична лимфоцитна левкемия разкрива понижаване на регулацията на задържаните интрони
Пълнометражни преписи|Секвениране на нанопори|Анализ на алтернативни изоформи
Заден план
Sшироко се съобщава, че оматичните мутации в фактора на сплайсинг SF3B1 се свързват с различни видове рак, включително хронична лимфоцитна левкемия (CLL), увеална меланома, рак на гърдата и т.н. В допълнение, краткотрайни транскриптомни изследвания разкриха аберантни модели на сплайсинг, индуцирани от мутации на SF3B1.Въпреки това, проучванията върху тези алтернативни модели на сплайсинг отдавна са ограничени до ниво на събитие и липса на знания на ниво изоформа поради ограничението на сглобените транскрипти за кратко четене.Тук беше въведена платформа за секвениране на нанопори за генериране на транскрипти с пълна дължина, което даде възможност за изследване на AS изоформи.
Експериментален дизайн
Експерименти
Групиране:1. CLL-SF3B1(WT) 2. CLL-SF3B1(K700E мутация);3. Нормални В-клетки
Стратегия за последователност:MinION 2D библиотечно секвениране, PromethION 1D библиотечно секвениране;данни за кратко четене от същите проби
Платформа за секвениране:ONT MinION;ONT PromethION;
Биоинформационен анализ
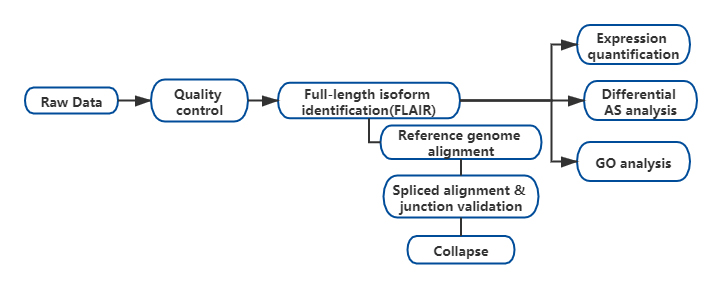
Резултати
Аобщо 257 милиона прочитания са генерирани от 6 CLL проби и 3 B-клетки.Средно 30,5% от тези четения са идентифицирани като преписи с пълна дължина.
Fалтернативен анализ на изоформа с пълна дължина на РНК (FLAIR) е разработен за генериране на набор от изоформи с висока степен на сигурност.FLAIR може да се обобщи като:
Nподравняване на четене на anopore: идентифициране на обща структура на транскрипт въз основа на референтен геном;
Sкорекция на plice съединение: коригирайте грешки в последователността (червено) с място на снаждане от анотирани интрони, интрони от кратко прочетени данни или и двете;
Collapse: обобщете представителни изоформи на базата на вериги за снаждане (набор за първо преминаване).Изберете isofrom с висока степен на сигурност въз основа на броя поддържащи четения (Праг: 3).
Фигура 1. FLAIR анализ за идентифициране на изоформи с пълна дължина, свързани с SF3B1 мутация в CLL
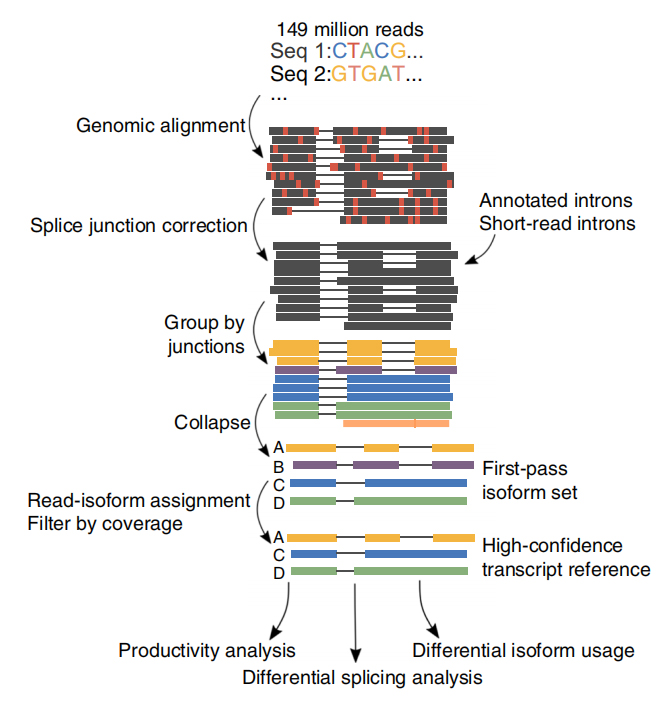
FLAIR Идентифицира 326 699 сплайсирани изоформи с висока степен на сигурност, 90% от които са нови изоформи.Установено е, че повечето от тези неанотирани изоформи са нови комбинации от известни снаждащи се съединения (142 971), докато останалите нови изоформи съдържат или запазен интрон (21 700), или нов екзон (3594).
Lнепрекъснато четени последователности позволяват идентифицирането на мутантни SF3B1-K700E -променени места на снаждане на ниво изоформа.Установено е, че 35 алтернативни 3'SS и 10 алтернативни 5'SS са значително различно свързани между SF3B1-K700E и SF3B1-WT.33 от 35-те промени са били новооткрити чрез отдавна прочетени последователности.В данните от Nanopore, разпределението на разстоянието между SF3B1-K700E-променени 3'SSs до пиковете на каноничните места е около -20 bp, което значително се различава от контролното разпределение, подобно на това, което е докладвано в CLL последователности за кратко четене.Изоформите на ERGIC3 гена бяха анализирани, където нова изоформа, съдържаща проксималното място на снаждане, беше намерена в по-голямо количество в SF3B1-K700E.Както проксималният, така и дисталният 3'SS бяха свързани с различни AS модели, генериращи множество изоформи.
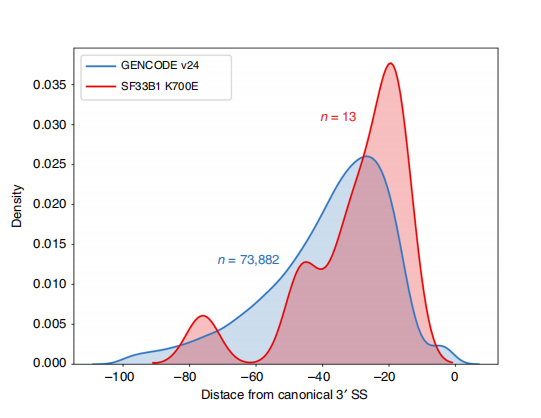
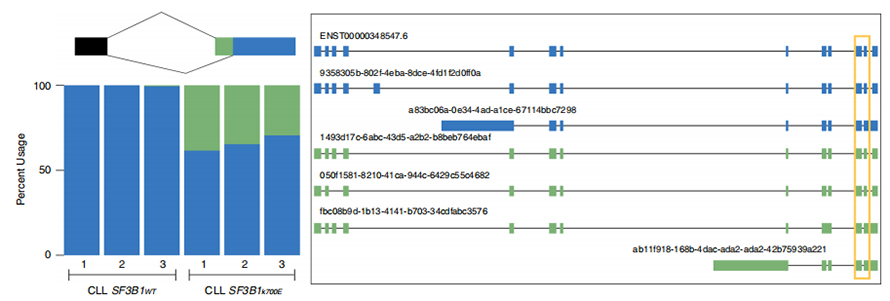
Фигура 2. Алтернативни 3' модели на снаждане, идентифицирани с данни за секвениране на нанопори
Анализът на използването на IR събития дълго време беше ограничен в анализа, базиран на кратко четене, поради доверието в идентифицирането и количественото определяне на IR.Експресията на IR изоформи в SF3B1-K700E и SF3B1-WT беше количествено определена въз основа на нанопорни последователности, разкривайки глобална низходяща регулация на IR изоформи в SF3B1-K700E.
Фигура 4. Интензивност на селското стопанство и мрежова свързаност в три земеделски системи (A и B);Случаен анализ на горите (C) и връзка между интензивността на земеделието и колонизацията на AMF (D)
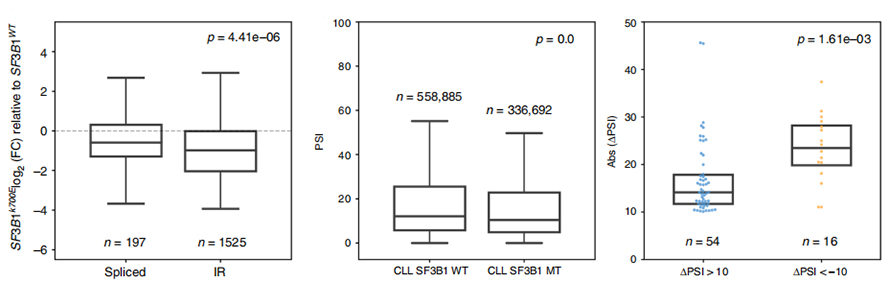
Фигура 3. Събитията за задържане на Intron са по-силно понижени в CLL SF3B1-K700E
технология
Секвениране на дълго четене на нанопори
Nанопорното секвениране е технология за секвениране на електрически сигнал в реално време с една молекула.
Dдвуверижната ДНК или РНК ще се свърже с нанопорест протеин, вграден в биофилма и ще се развива под ръководството на моторен протеин.
DNA/RNA нишките преминават през протеин на нанопорен канал с определена скорост под действието на разликата в напрежението.
Mмолекулите генерират различни електрически сигнали според химичната структура.
Rоткриването на последователностите във всеки момент се постига чрез извикване на база.
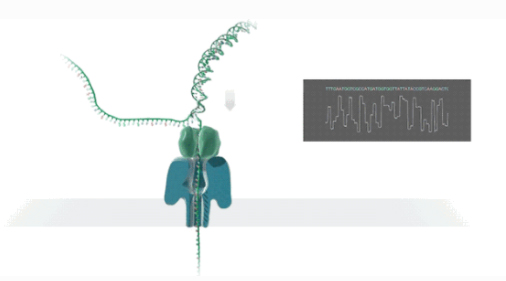
Изпълнение на секвениране на транскриптоми в пълна дължина
√ Наситеност на данни
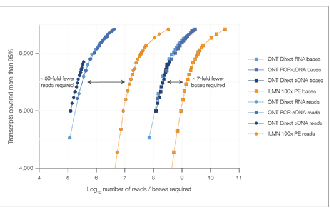
Необходими са 7 пъти по-малко четения за достигане на сравнимо насищане на данни.
√ Идентификация на структурата на транскрипта
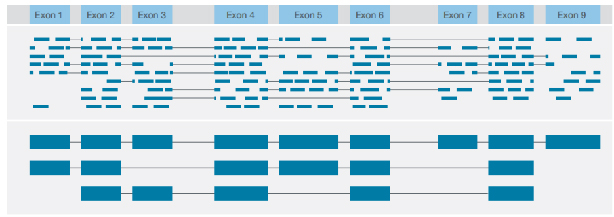
Идентифициране на различни структурни варианти с консенсусно прочитане на цяла дължина на всеки транскрипт
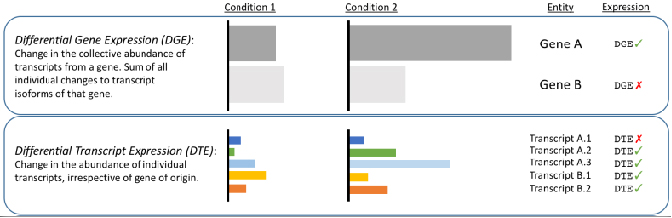
√ Диференциален анализ на ниво транскрипт - Разкриване на промени, скрити от кратко четене
справка
Tang AD, Soulette CM, Baren MJV и др.Характеризиране на препис с пълна дължина на SF3B1 мутация при хронична лимфоцитна левкемия разкрива понижаване на регулацията на задържаните интрони [J].Nature Communications.
Технологии и акценти има за цел да сподели най-новото успешно приложение на различни технологии за секвениране с висока производителност в различни изследователски среди, както и брилянтни идеи в експерименталния дизайн и извличането на данни.
Време на публикуване: 8 януари 2022 г

